9.Biomarker
Aufgabe 1: Definitionen
ROC: Abkürzung für Receiver Operating Characteristic. Dies ist eine Methode mit der man Analysemethoden wie z.B. Diagnosealgorithmen optimieren und bewerten kann. Dafür bestimmt man die falschpositiven und falschnegativen Ergebnisse der Analysemethode und bestimmt damit die Sensitivität und Spezifität der Methode. Anschließend wägt man diese gegeneinander ab. Dies geschieht mittels der Grenzwertoptimierungskurve (ROC- Kurve).
AUC: Abkürzung für Area under Curve. Beschreibt die Fläche unter der Kurve die man erhält wenn man Sensitivität gegen 1-Spezifität aufträgt. Der AUC-Wert liegt dabei zwischen 1 und 0. Ein Wert nahe 1 bedeutet dabei dass die Analysemethode wenig falschpositive und falschnegative Werte generiert. Ein Wert nahe 0 bedeutet dass die Analysemethode fast ausschließlich falschpositive und falschnegative Werte generiert. Dies bedeutet, dass der Algothitmus trotzdem funktioniert, man ihn jedoch "umdrehen" muss. Werte um 0,5 herum sind daher die schlechtesten Werte, da diese bedeuten dass die Methode qualitativ so gut sind wie per Zufall zu diagnositizieren.
Spezifität: [math]\displaystyle{ \frac{True Negatives}{(False Positives+True Negatives)} }[/math]
Rate, mit denen die Analysemethode Negativproben korrekt erkennt werden. Idealerweise geht der Werte gegen 1.
Sensitivität: [math]\displaystyle{ \frac{True Positives}{(False Negatives+True Positives)} }[/math]
Rate, mit denen die Analysemethode Positivproben korrekt erkennt werden. Idealerweise geht der Werte gegen 1.
logit-Funktion: eine logistische Funktion, die Analysemethoden verwenden, um mit Expressionsleveln von Biomarkern einen Wert zwischen 0 und 1 zu berechnen. Dieser kann dann verwendet werden, um abzuschätzen, ob ein positives oder ein negatives Ergebnis vorliegt.
[math]\displaystyle{ logit(P) = log \frac{P_{X}}{1-P_{X}} = \beta_{1} * TPM_{GenX} + \beta_{0} }[/math]
(Zähler=Wahrscheinlichkeit; Nenner=Gegenwahrscheinlichkeit)
Threshold: Cutoff-Wert in der logit-Funktion. Dort trennt der Analysealgrithmus die logit-Funktion.
Alles auf einer Seite wird als positiv bewertet und alles auf der anderen Seite als negativ. Ob positiv krank bedeutet, hängt davon ab, ob bei kranken Menschen dieser Biomarker hoch- oder runtereguliert ist.
Anderer Erklärungsansatz:
- Ein Grenzwert zwischen 2 Konditionen
- Wird anhand Trainingsdaten justiert
- Beispiel: krank=1; gesund=0; Threshold=0.5; Alle Werte unter 0.5 sind gesund
Aufgabe 2: Biomarker
a: Was ist ein Biomarker und wofür kann er verwendet werden?
Biomarker sind messbare biologische Parameter die eine gewisse Aussagekraft für Diagnose und Prognose haben. Mit der Analyse von Biomarkern kann man bei der Diagnose von Krankheiten bei Menschen helfen. Das Protein Troponin T ist beispielweise der Indikator in der Herzinfarkdiagnostik. Da dieses Protein in Herzmuskeln besonders exprimirt wird und als Konsequenz eines sterbenden Herzmuskels in die Blutbahn gelangt, wird dieser Wert bei Brustbeschwerden als joch signifikant für die weitere Diagnostik oder Therapie angesehen. Ein moderner Ansatz in der Bioinformatik wäre bspw. die Genexpression als Biomarker zu verwenden. Möglicherweise könnten so Tumorzellen sicher im Blut detektiert werden, da sich ihre Genexpression von physiologische Zellen deutlich unterscheidet. Eine standardisierte, klinische Anwendung ist bisher jedoch nicht möglich.
b: Beschreiben Sie, wie Biomarker bestimmt werden können.
Einfache Biomarker wie Körpertemperatur und Blutdruck kann man mit einfachen diagnostischen Methoden bestimmen (Fieberthermometer bzw Oberarm-Manometer).
Um Genexpression als Biomarker zu nutzen, kann mit einer RNA-Sequenzierung eine 'verdächtige' Probe mit einer Referenzprobe vergleichen. Die Werte kann man anschließend mit Analysemethoden aufbereiten, und am Besten mit mehreren Biomarkern eine zuverlässigere Vorraussage treffen.
Grundsätzlich liefert die Bioinformatik hier viele Möglichkeiten neue Biomarker in der medizinischen Diagnostik langfristig zu etablieren:
- Klare Unterschiede zwischen zu untersuchenden Zuständen erkennbar (Bspw: Metabolitlevel - Expressionslevel)
- Auswertung großer Datensätze mit bioinformatischen Methoden wie 'machine learning'
- Auf den Patienten hochgradig zugeschnittene Behandlungskonzepte im Sinne von 'individualized medicine'
c: Warum ist ein einzelner Biomarker meistens nicht genug und was ist der Vorteil einer Biomarker-Signatur?
Ein Biomarker reicht oft nicht, da die Werte mehr oder weniger um einen Mittelwert streuen. Ein Wert kann also durchaus mal aus Ausreißer jenseits des Thresholdes liegen und ein 'falsch positives' oder 'falsch negatives' Ergebnis erzeugen. Die Signifikanz eines Testes kann durch die Verwendung mehrerer mit der Krankheit assoziierter Biomarker (gennant Biomarker-Signatur) deutlich erhöht werden. Dies erhöht also sowohl die Spezifität als auch die Sensivität.
Aufgabe 3
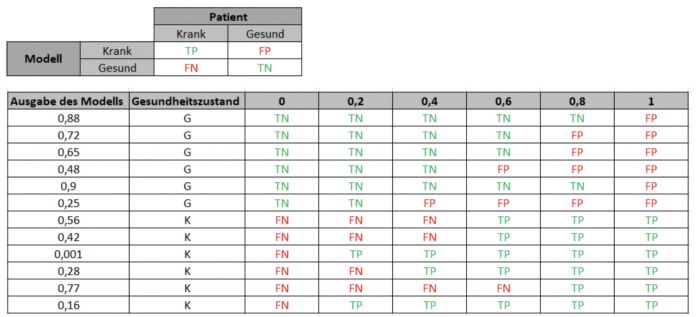
Aus den ermittelten Metabolitleveln einer imaginären Studie wurde ein logistisches Regressionsmodell entworfen. Dieses Modell soll in der Lage sein, an Hand von diesen Metabolitleveln eine Vorhersage über den Gesundheitszustand von Patienten zu machen. Bei dem Aufstellen des Modells wurde eine 0 als krank und eine 1 als gesund definiert. Folgende Ausgaben aus diesem Modell wurden enthalten:
Bitte bearbeiten Sie nun folgende Fragestellungen:
a: Was sind FPR, TPR, TNR und FNR? Erklären Sie kurz!
- FPR: False Positive Rate: Rate an Patienten, die gesund sind, aber vom Modell als krank erachtet werden.
- TPR: True Positive Rate: Rate an Patienten, die krank sind, und vom Modell als krank erachtet werden.
- TNR: True Negative Rate: Rate an Patienten, die gesund sind, und vom Modell als gesund erachtet werden.
- FNR: False Negative Rate: Rate an Patienten, die krank sind, aber vom Modell als gesund erachtet werden.
TPR = Sensivität
TNR = Spezifität
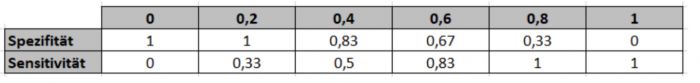
b) Berechnen Sie für die 6 Thresholds 0; 0,2; 0,4; 0,6; 0,8; 1 die Spezifität und die
Sensitivität des Modells.
c) Welcher dieser Thresholds ist für dieses Modell der Beste? Begründen Sie!
Grundsätzlich sollte ein Threshold gewählt werden, bei dem Sensivität und Spezifität möglichst gegen 1 gehen. Hier besitzt bspw der Threshold von 0.6 den höchsten Wert für die Summe aus Spezifität und Sensivität.
Bei vielen Modellen muss jedoch entschieden werden, ob der Test eher sensibel oder spezifisch sein soll. Die passende Threshold hängt also davon ab, welche Krankheit man detektieren möchte. Je schwerwiegender diese ist oder je einfacher die Diagnose nach einem Treffer mit dem Modell, desto eher kann auch eine geringe Spezifität für eine möglichst hohe Sensitivität in Kauf nehmen.