3 Alignments
Sequenzalignment
Das optimale „Aneinander ausrichten“ von Sequenzen, sodass z.B. reads aus einer Sequenzierung an ein Referenzgenom ausgerichtet werden können.
Es gibt enorm viele Möglichkeiten eine Sequenz an eine andere zu Assemblieren. Deshalb benötigt man speziell Algorithmen, die die besten Möglichkeiten finden. Um zu bestimmen welches Alignment das beste ist, wird ein Bewertungsschema, der sogenannte Score, angewandt. So kann die Übereinstimmung zweier Sequenzen miteinander quantitativ (mit Zahlen) verglichen werden. Zudem ermöglicht das Sequenzalignment die Erkennung ähnlicher Domänen und erfordert das Einfügen von gaps (s. Smith-Waterman).
Man unterscheidet zwei verschiedenen Arten beim Sequenzalignment:
Globales Alignment
- Alignment zwischen zwei Sequenzen, die ähnlich lang sind und bei denen starke Sequenzhomologien erwartet werden
- Vergleich von Gesamtsequenzen
- alle Symbole werden berücksichtigt
- zur Berechnung des optimalen Alignment Scores wird häufig der Needle-Wunsch Algorithmus benutzt
Beispiel:
| H | A | U | S | K | L | A | U | S | ||
| ↓ | ||||||||||
| H | - | A | U | S | - | H | A | U | S | |
| K | L | A | U | S | K | L | A | U | S | |
| Score 8 | Score 8 | |||||||||
Bewertungsschema:
S(a,b)={3 a=b match ;0 a≠b mismatch
| gaps: | S(a,-)=-1 Deletion |
| S(-,b)=-1 Insertion |
Lokales Alignment
- Alignment von Teilsequenzen
- Vergleich zweier sehr unterschiedlicher Sequenzen, die aber gleiche Motive besitzen
- z.B. die Suche einer Gensequenz im Genom
Beispiel:
| A | T | G | C | A | T | T | A | C |
| C | T | T | T | A |
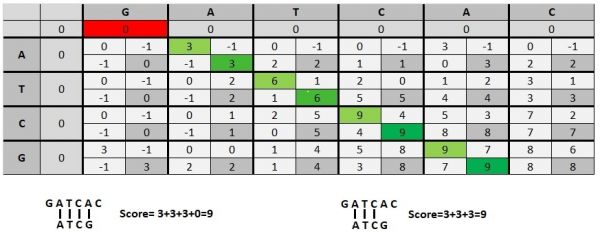
Smith-Waterman Algorithmus
Dynamic programming: "divide and conquer", Aufteilen des Problems in Subprobleme
M0,0=0
M0,j=0
Mk,0=0
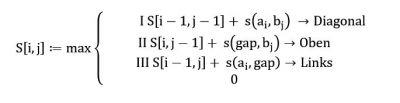
Das Alignment beginnt bei dem höchsten erzielten Score in der Matrix
Score: Match: +3 | Mismatch: 0 | Gap: -1