10.BLAST: Difference between revisions
No edit summary |
No edit summary |
||
| Line 39: | Line 39: | ||
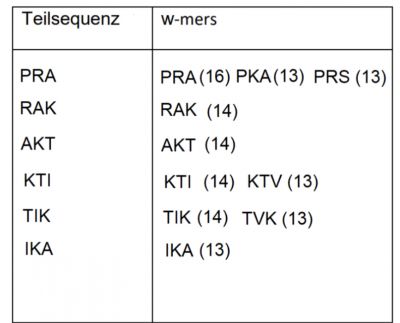
'''d''': Bestimme manuell für die Sequenz 'PRAKTIKA' sämtliche w-mers der Länge 3, die bei Verwendung der BLOSUM62 Matrix einen Score T>13 besitzen. | '''d''': Bestimme manuell für die Sequenz 'PRAKTIKA' sämtliche w-mers der Länge 3, die bei Verwendung der BLOSUM62 Matrix einen Score T>13 besitzen. | ||
[[File:BLAST1.jpeg| | [[File:BLAST1.jpeg|400px|center|thumb]]<br /> | ||
Revision as of 18:56, 4 October 2020
Grundlagen
a: Was ist BLAST?
- Basic local alignment search tool
- Schnelle Suche einer Sequenz in einer Sequenzdatenbank
- Sequenz wird zur Suche in Teilsequenzen zerlegt
- kann zur Strukur- und Funktionsvorhersage dienen
b: Auf welchem Prinzip beruht BLAST? Erkläre die Funktionsweise.
Er sucht nach homologen Sequenzen in der Datenbank und berechnet zusätzlich eine statistische Signifikanz
Die Idee des Algorithmus basiert auf der Wahrscheinlichkeit, dass Alignment mit vielen Treffern auch viele zufällig ausgewählte Stücke von großerer Identität (im Sinne von identisch) besitzen. Diese gefunden Teilstücke werden dann während der Suche nach besseren und längeren Alignments weiter vergrößert. Indem diese Segmente kurz gehalten werden, ist es möglich, die Abfragesequenz vor einer Suche zu bearbeiten. Dadurch kann eine Tabelle mit möglichen Teilstücken mit ihrem Ursprung in der Orginalsequenz vorgehalten werden.
Dabei stellt der Algorithmus eine Liste aller benachbarten Worte fester Länge auf, die einen Treffer auf der Abfragesequenz mit einem höheren Scoring als ein zu wählender Parameter erzeugen würden. Anschließend wird die Zieldatenbank nach Wörtern in dieser Liste abgefragt und die gefundenen Treffer erweitert, um mögliche maximal zusammenhängende Treffer -in beide Richtungen- zu finden.
Der Ablauf dafür ist:
- 1. Die 'query' Sequenz wird in Worte der Länge 'l' zerlegt (l = 3 für Proteine; l = 11 für DNA)
- 2. Für jedes Wort wird eine Liste ähnlicher Wörter (w-mers) mit Score S>Threshold erzeugt (Threshold=13 bei Aminosäuresequenzen; basierend auf der BLOSUM62 Matrix)
- 3. Alle w-mers der Liste werden in der Datenbank gesucht und die Position bestimmt
- 4. Die Treffer werden erweitert, bis der Score kleiner wird
- 5. Ist der Score größer als der cutoff, handelt es sich um ein 'high-scoring-pair' (HSP)
- 6. HSPs werden durch lokales Alignment erweiter (Smith Waterman), der echte Alignment Score wird berechnet. Gaps können nun berücksichtig werden.
c: Um was für einen Algorithmus handelt es sich bei BLAST? Nenne einen Vor- und einen Nachteil.
- heuristischer Algorithmus
Vorteil: schnell Nachteil: nichts zwangsläufig das beste Alignment wird gefunden, abhängig von den Eingangsparametern; kann Fusionproteine nicht finden
d: Bestimme manuell für die Sequenz 'PRAKTIKA' sämtliche w-mers der Länge 3, die bei Verwendung der BLOSUM62 Matrix einen Score T>13 besitzen.