6 Normalisierungen: Difference between revisions
No edit summary |
No edit summary |
||
| Line 4: | Line 4: | ||
Normalisierungen der RNASeq-Daten sind notwendig, um diese vergleichbar machen zu können und die tatsächliche Genexpression quantifizieren zu können. | Normalisierungen der RNASeq-Daten sind notwendig, um diese vergleichbar machen zu können und die tatsächliche Genexpression quantifizieren zu können. | ||
==RPKM== | ==RPKM== | ||
RPKM steht für 'Reads per kilobase of transcript per Million mapped reads'. <br> | RPKM steht für 'Reads per kilobase of transcript per Million mapped reads' und ist eine Normalisierungsmethode für Expressionsstärken. <br> | ||
{| class="wikitable" | {| class="wikitable" | ||
|- | |- | ||
| Line 10: | Line 10: | ||
|- | |- | ||
| <math> RPKM = \frac{ c_\text{i}}{L_\text{i} \cdot N} </math> || c<sub>i</sub> = Anzahl an ausrichtbaren reads für ein Transkript 'i'<br> | | <math> RPKM = \frac{ c_\text{i}}{L_\text{i} \cdot N} </math> || c<sub>i</sub> = Anzahl an ausrichtbaren reads für ein Transkript 'i'<br> | ||
L<sub>i</sub> = Länge des Transkripts/Gens 'i' in | L<sub>i</sub> = Länge des Transkripts/Gens 'i' in kbp <br> | ||
N = Gesamtanzahl an ausrichtbaren reads in Millionen | N = Gesamtanzahl an ausrichtbaren reads in Millionen | ||
|} <br> | |} | ||
<math> \frac{ c_\text{i}}{L_\text{i}} </math> → Normalisierung der Genlänge <br> | |||
<math> L_\text{i} \cdot N </math> → Normalisierung der Sequenziertiefe <br> | |||
Je höher der RPKM-Wert ist, desto höher ist die Genexpression. <br> | |||
RPKM-Werte können gut zum Vergleich der Genexpressionen innerhalb einer Probe genutzt werden, jedoch sind sie nicht dafür geeignet mehrere Experimente bzw. Proben miteinander zu vergleichen. | |||
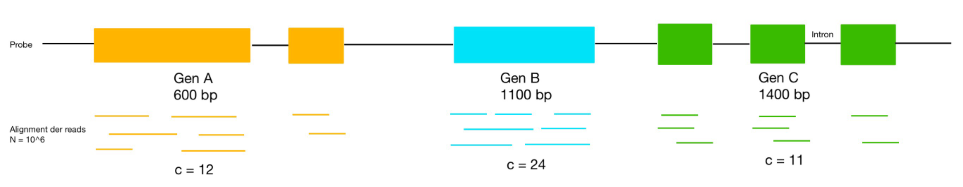
'''Beispiel:''' | |||
[[File:Beispiel Probe.png|Beispiel Probe]] <br><br> | [[File:Beispiel Probe.png|Beispiel Probe]] <br><br> | ||
* <math> RPKM (A) = \frac{12}{0,6 kbp \cdot 6} = 3,33 </math> <br> | |||
* <math> RPKM (B) = \frac{24}{1,1 kbp \cdot 6} = 3,64 </math> <br> | |||
* <math> RPKM (C) = \frac{12}{1,4 kbp \cdot 6} = 1,43 </math> <br><br> | |||
Durch die Normalisierung der Expressionsstärken können diese nun miteinander vergleichen werden.In diesem Beispiel wird Gen B am stärksten exprimiert und Gen C am schwächsten. | |||
==TPM== | ==TPM== | ||
TPM steht für 'Transcripts per Million'. <br> | TPM steht für 'Transcripts per Million' und ist eine Normalisierungsmethode für Expressionsstärken. <br> | ||
{| class="wikitable" | {| class="wikitable" | ||
| Line 27: | Line 36: | ||
|- | |- | ||
|<math> TPM = \frac{ c_\text{i}}{L_\text{i}} \cdot \dfrac {1}{\sum_\text{n} \dfrac {c_\text{n}}{L_\text{n}}} \cdot 10^6 </math> <br> || c<sub>i</sub> = Anzahl an ausrichtbaren reads für ein Transkript 'i'<br> | |<math> TPM = \frac{ c_\text{i}}{L_\text{i}} \cdot \dfrac {1}{\sum_\text{n} \dfrac {c_\text{n}}{L_\text{n}}} \cdot 10^6 </math> <br> || c<sub>i</sub> = Anzahl an ausrichtbaren reads für ein Transkript 'i'<br> | ||
L<sub>i</sub> = Länge des Transkripts/Gens 'i' in | L<sub>i</sub> = Länge des Transkripts/Gens 'i' in kbp <br> | ||
N = Gesamtanzahl an ausrichtbaren reads in Millionen | N = Gesamtanzahl an ausrichtbaren reads in Millionen | ||
|} <br> | |} <br> | ||
Revision as of 18:02, 3 June 2019
Auf dieser Seite sind die Themen zusammengeführt, die in Vorlesung 5 am 16.05.2019 behandelt wurden.
Normalisierungen
Normalisierungen der RNASeq-Daten sind notwendig, um diese vergleichbar machen zu können und die tatsächliche Genexpression quantifizieren zu können.
RPKM
RPKM steht für 'Reads per kilobase of transcript per Million mapped reads' und ist eine Normalisierungsmethode für Expressionsstärken.
| Formel | Parameter |
|---|---|
| [math]\displaystyle{ RPKM = \frac{ c_\text{i}}{L_\text{i} \cdot N} }[/math] | ci = Anzahl an ausrichtbaren reads für ein Transkript 'i' Li = Länge des Transkripts/Gens 'i' in kbp |
[math]\displaystyle{ \frac{ c_\text{i}}{L_\text{i}} }[/math] → Normalisierung der Genlänge
[math]\displaystyle{ L_\text{i} \cdot N }[/math] → Normalisierung der Sequenziertiefe
Je höher der RPKM-Wert ist, desto höher ist die Genexpression.
RPKM-Werte können gut zum Vergleich der Genexpressionen innerhalb einer Probe genutzt werden, jedoch sind sie nicht dafür geeignet mehrere Experimente bzw. Proben miteinander zu vergleichen.
Beispiel:
- [math]\displaystyle{ RPKM (A) = \frac{12}{0,6 kbp \cdot 6} = 3,33 }[/math]
- [math]\displaystyle{ RPKM (B) = \frac{24}{1,1 kbp \cdot 6} = 3,64 }[/math]
- [math]\displaystyle{ RPKM (C) = \frac{12}{1,4 kbp \cdot 6} = 1,43 }[/math]
Durch die Normalisierung der Expressionsstärken können diese nun miteinander vergleichen werden.In diesem Beispiel wird Gen B am stärksten exprimiert und Gen C am schwächsten.
TPM
TPM steht für 'Transcripts per Million' und ist eine Normalisierungsmethode für Expressionsstärken.
| Formel | Parameter |
|---|---|
| [math]\displaystyle{ TPM = \frac{ c_\text{i}}{L_\text{i}} \cdot \dfrac {1}{\sum_\text{n} \dfrac {c_\text{n}}{L_\text{n}}} \cdot 10^6 }[/math] |
ci = Anzahl an ausrichtbaren reads für ein Transkript 'i' Li = Länge des Transkripts/Gens 'i' in kbp |
TMM
TMM steht für 'Trimmed mean of M-values normalization method'.
[math]\displaystyle{ M_\text{gk}^\text{k`} = log_2 \dfrac { \dfrac {Y_\text{gk}}{N_\text{k}}}{ \dfrac {Y_\text{gk`}}{N_\text{k`}}} }[/math]
[math]\displaystyle{ log_2 (TMM_\text{k}^\text{k`}) = \frac{ \underset {g \in G}{\sum} W_\text{gk}^\text{k´} \cdot M_\text{gk}^\text{k´}} { \sum_{g \in G} W_\text{gk}^\text{k´}} }[/math]
[math]\displaystyle{ W_\text{gk}^\text{k`} = \frac{ N_\text{k} - Y_\text{gk}}{N_\text{k} \cdot Y_\text{gk}} + \frac{ N_\text{k`} - Y_\text{gk`}}{N_\text{k`} \cdot Y_\text{gk`}} }[/math]
Der Spaß hier wird morgen erweitert, hab mich nur mit den Formeln vertraut gemacht :D _ Vero