6 Normalisierungen: Difference between revisions
No edit summary |
No edit summary |
||
| Line 4: | Line 4: | ||
Normalisierungen der RNASeq-Daten sind notwendig, um diese vergleichbar machen zu können und die tatsächliche Genexpression quantifizieren zu können. | Normalisierungen der RNASeq-Daten sind notwendig, um diese vergleichbar machen zu können und die tatsächliche Genexpression quantifizieren zu können. | ||
==RPKM== | ==RPKM== | ||
RPKM steht für 'Reads per kilobase of transcript per Million mapped reads' <br> | RPKM steht für 'Reads per kilobase of transcript per Million mapped reads'. <br> | ||
<math> RPKM = \frac{ c_\text{i}}{L_\text{i} \cdot N} </math> | {| class="wikitable" | ||
|- | |||
! Formel !! Parameter | |||
|- | |||
| <math> RPKM = \frac{ c_\text{i}}{L_\text{i} \cdot N} </math> || c<sub>i</sub> = Anzahl an ausrichtbaren reads für ein Transkript 'i'<br> | |||
L<sub>i</sub> = Länge des Transkripts/Gens 'i' in Kbp <br> | |||
N = Gesamtanzahl an ausrichtbaren reads in Millionen | |||
|} <br> | |||
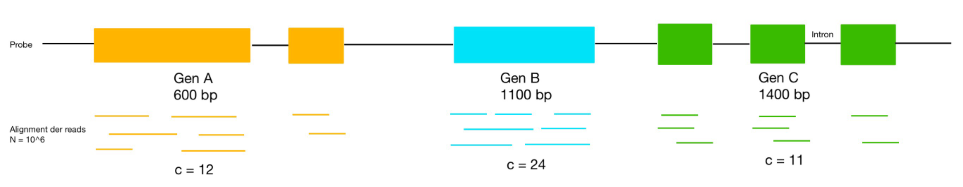
Beispiel: <br> | |||
[[File:Beispiel Probe.png|Beispiel Probe]] <br><br> | |||
==TPM== | ==TPM== | ||
TPM steht für 'Transcripts per Million' <br> | TPM steht für 'Transcripts per Million'. <br> | ||
<math> TPM = \frac{ c_\text{i}}{L_\text{i}} \cdot \dfrac {1}{\sum_\text{n} \dfrac {c_\text{n}}{L_\text{n}}} \cdot 10^6 </math> | |||
{| class="wikitable" | |||
|- | |||
! Formel !! Parameter | |||
|- | |||
|<math> TPM = \frac{ c_\text{i}}{L_\text{i}} \cdot \dfrac {1}{\sum_\text{n} \dfrac {c_\text{n}}{L_\text{n}}} \cdot 10^6 </math> <br> || c<sub>i</sub> = Anzahl an ausrichtbaren reads für ein Transkript 'i'<br> | |||
L<sub>i</sub> = Länge des Transkripts/Gens 'i' in Kbp <br> | |||
N = Gesamtanzahl an ausrichtbaren reads in Millionen | |||
|} <br> | |||
==TMM== | ==TMM== | ||
TMM steht für 'Trimmed mean of M-values normalization method' | TMM steht für 'Trimmed mean of M-values normalization method'. | ||
<math> M_\text{gk}^\text{k`} = log_2 \dfrac { \dfrac {Y_\text{gk}}{N_\text{k}}}{ \dfrac {Y_\text{gk`}}{N_\text{k`}}} </math> | <math> M_\text{gk}^\text{k`} = log_2 \dfrac { \dfrac {Y_\text{gk}}{N_\text{k}}}{ \dfrac {Y_\text{gk`}}{N_\text{k`}}} </math> | ||
Revision as of 17:23, 3 June 2019
Auf dieser Seite sind die Themen zusammengeführt, die in Vorlesung 5 am 16.05.2019 behandelt wurden.
Normalisierungen
Normalisierungen der RNASeq-Daten sind notwendig, um diese vergleichbar machen zu können und die tatsächliche Genexpression quantifizieren zu können.
RPKM
RPKM steht für 'Reads per kilobase of transcript per Million mapped reads'.
| Formel | Parameter |
|---|---|
| [math]\displaystyle{ RPKM = \frac{ c_\text{i}}{L_\text{i} \cdot N} }[/math] | ci = Anzahl an ausrichtbaren reads für ein Transkript 'i' Li = Länge des Transkripts/Gens 'i' in Kbp |
Beispiel:
TPM
TPM steht für 'Transcripts per Million'.
| Formel | Parameter |
|---|---|
| [math]\displaystyle{ TPM = \frac{ c_\text{i}}{L_\text{i}} \cdot \dfrac {1}{\sum_\text{n} \dfrac {c_\text{n}}{L_\text{n}}} \cdot 10^6 }[/math] |
ci = Anzahl an ausrichtbaren reads für ein Transkript 'i' Li = Länge des Transkripts/Gens 'i' in Kbp |
TMM
TMM steht für 'Trimmed mean of M-values normalization method'.
[math]\displaystyle{ M_\text{gk}^\text{k`} = log_2 \dfrac { \dfrac {Y_\text{gk}}{N_\text{k}}}{ \dfrac {Y_\text{gk`}}{N_\text{k`}}} }[/math]
[math]\displaystyle{ log_2 (TMM_\text{k}^\text{k`}) = \frac{ \underset {g \in G}{\sum} W_\text{gk}^\text{k´} \cdot M_\text{gk}^\text{k´}} { \sum_{g \in G} W_\text{gk}^\text{k´}} }[/math]
[math]\displaystyle{ W_\text{gk}^\text{k`} = \frac{ N_\text{k} - Y_\text{gk}}{N_\text{k} \cdot Y_\text{gk}} + \frac{ N_\text{k`} - Y_\text{gk`}}{N_\text{k`} \cdot Y_\text{gk`}} }[/math]
Der Spaß hier wird morgen erweitert, hab mich nur mit den Formeln vertraut gemacht :D _ Vero