5 Transkriptom RNA Seq 2: Difference between revisions
From Bioinformatik Wiki
| Line 58: | Line 58: | ||
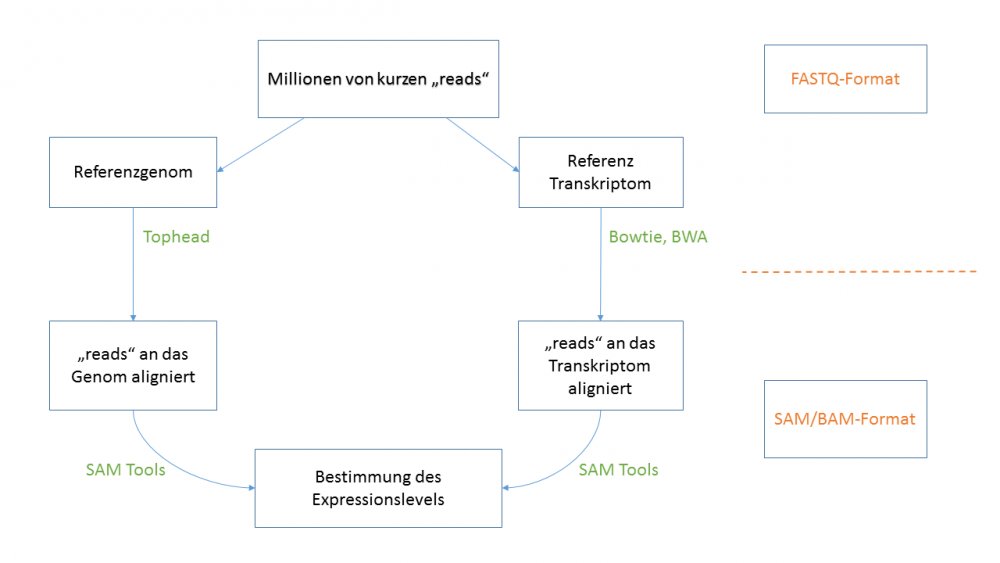
== 5. Read Alignment == | == 5. Read Alignment == | ||
[[File:Read-alignment.png|center|1000px]] | [[File:Read-alignment.png|center|1000px]] | ||
=== SAM und BAM Format === | |||
* Enthält Informationen über die Sequenz-reads nach dem Alignment an ein Referenzgenom. | |||
* SAM= Sequence Alignment/Map | |||
* BAM= Binary Alignment/Map | |||
* SAM ist ein textbasiertes Dateiformat und BAM ein binäres Dateiformat. | |||
Revision as of 15:30, 27 September 2021
RNAseq
RNA sequencing ist eine Methode zur Analyse von Transkriptomen. Hierbei wird die Sequenz aller in der Probe vorhandenen mRNA Moleküle durch Next-Generation Sequencing (NGS) ermittelt.
1. Isolierung von mRNA
- nur 1-2% der totalen RNA ist mRNA
- 90% rRNA
- weil wir nur die mRNA isolieren wollen, müssen wir diese von der restlichen RNA trennen
- das ist entweder durch die sogennante poly(A) Anreicherung möglich, dabeimacht man sich die besondere Eigenschaft der mRNA zu nutze
- diese besitzt im Gegensatz zu anderen RNA Spezies einen polyA-Schwanz, immobilisiert man auf der Matrix einer Chromatofraphiesäule polyT-Seqeunezen, kann die mRNA komplementär an diese binden und kann so hufgereinigt werden
- die zweite Möglichkeit ist der spezifische Abbau von rRNA
- diese Vorgehensweise gewinnt vor allem bei Prokaryotischer mRNA an Bedeutung, bei der es keinen polyA-Schwanz gibt
2. cDNA Synthese und Library Präparation
- Information über kodierenden Strang kann durch 2-stufige cDNA Synthese und dUTP Nukleotiden konserviert werden.
- Abbau des non-coding Stranges durch durch Uracil-DNA-Glykosylase
- baut spezifisch den non-coding Strang ab, weil dieser als einziger Uracil besitzt
- d.h. alle zum cDNA-Originalstrang komplementären Stränge werden abgebaut
- durch die Adapter bleibt die Topologie erhalten
- es folgt die Amplifikation mittels PCR
3. Sequenzierung/ Next generation Sequencing
- Sequenziertiefe bestimmt die Empfindlichkeit und die Genauigkeit
- 5 Millionen reads ausreichend für mittel-hoch exprimierte Gene
- 100 Millionen reads für schwach exprimierte Gene
- wichtig: Anzahl an Replikaten bestimmt statistische Sensitivität für Unterschiede
| Replikate pro Gruppe | |||
| fold change | 3 | 5 | 10 |
| 1,25 | 17% | 25% | 44% |
| 1,5 | 43% | 64% | 91% |
| 2 | 87% | 98% | 100% |
4. Datenanalyse
Präsprozessierung der Rohdaten:
⇒Filtern von Basen mit geringer Sequenzierqualität
⇒ Trimmen von Adaptersequenzen und PCR-Primersequenzen
⇒ Programme: FASTQC, NGSQC, Trimmomatic
5. Read Alignment
SAM und BAM Format
- Enthält Informationen über die Sequenz-reads nach dem Alignment an ein Referenzgenom.
- SAM= Sequence Alignment/Map
- BAM= Binary Alignment/Map
- SAM ist ein textbasiertes Dateiformat und BAM ein binäres Dateiformat.