3 Alignments: Difference between revisions
| (10 intermediate revisions by 2 users not shown) | |||
| Line 1: | Line 1: | ||
= | = Sequenzalignment = | ||
Das optimale „Aneinander ausrichten“ von Sequenzen, sodass z.B. reads aus einer Sequenzierung an ein Referenzgenom ausgerichtet werden können. | Das optimale „Aneinander ausrichten“ von Sequenzen, sodass z.B. reads aus einer Sequenzierung an ein Referenzgenom ausgerichtet werden können. <br> | ||
Es gibt enorm viele Möglichkeiten eine Sequenz an eine andere zu alignen. Deshalb benötigt man spezielle Algorithmen, die die besten Möglichkeiten finden. Um zu bestimmen welches Alignment das beste ist, wird ein Bewertungsschema, der sogenannte alignment score, angewandt. So kann die Übereinstimmung zweier Sequenzen miteinander quantitativ (mit Zahlen) verglichen werden. <br> | |||
Es werden nicht alle möglichen Alignments ausprobiert und dann deren Scores verglichen, um herauszufinden welches Alignment das beste ist. Mit dieser "brute force" Methode ergeben sich bei kurzen Sequenzen von 100 Basen schon 10<sup><big>75</big></sup> Alignmentmögichkeiten. Stattdessen werden Sequenzen mit Hilfe des "Dynamic Programming" alignt. Es folgt dem Prinzip von "divide and conquer", also die Zerlegung des Problems in viele Unterprobleme, die nacheinander gelöst werden. | |||
Zudem ermöglicht das Sequenzalignment die Erkennung ähnlicher Domänen. Da oft sogar homologe Sequenzen, die von einer gemeinsamen Sequenz abstammen, durch indels, die im Laufe der Evolution aufgetreten sind, unterschiedliche Längen besitzen, ist es notwendig gaps Einzufügen um die Ähnlichkeit der beiden Sequenzen sichtbar zu machen (s. Smith-Waterman). | |||
Anwenndungen finden Alignments im Sequenzvergeleich, z.B. bei phylogenetischen Untersuchungen. | |||
Man unterscheidet zwei verschiedenen Arten beim Sequenzalignment: | |||
== Globales Alignment == | == Globales Alignment == | ||
* | * Alignment zwischen zwei Sequenzen, die ähnlich lang sind und bei denen starke Sequenzhomologien erwartet werden | ||
* Vergleich von Gesamtsequenzen | |||
* alle Symbole werden berücksichtigt | * alle Symbole werden berücksichtigt | ||
* | * zur Berechnung des optimalen Alignment Scores wird häufig der Needle-Wunsch Algorithmus benutzt | ||
<br> | <br> | ||
'''Beispiel''':<br> | '''Beispiel''':<br> | ||
| Line 45: | Line 46: | ||
* Alignment von Teilsequenzen | * Alignment von Teilsequenzen | ||
* Vergleich zweier sehr unterschiedlicher Sequenzen, die aber gleiche Motive besitzen | * Vergleich zweier sehr unterschiedlicher Sequenzen, die aber gleiche Motive besitzen | ||
*Suche einer Gensequenz im Genom | * z.B. die Suche einer Gensequenz im Genom | ||
<br> | <br> | ||
Beispiel:<br> | Beispiel:<br> | ||
| Line 56: | Line 57: | ||
|} | |} | ||
</big> | </big> | ||
==Smith-Waterman Algorithmus== | |||
Dynamic programming: "divide and conquer", Aufteilen des Problems in Subprobleme | Dynamic programming: "divide and conquer", Aufteilen des Problems in Subprobleme | ||
<br> | <br> | ||
| Line 64: | Line 65: | ||
M<sub>0,j</sub>=0<br> | M<sub>0,j</sub>=0<br> | ||
M<sub>k,0</sub>=0<br> | M<sub>k,0</sub>=0<br> | ||
[[File:Smith Waterman Algorithmus.jpg|400px|frameless]] | |||
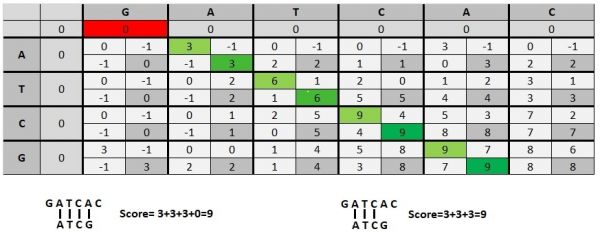
Das Alignment beginnt bei dem höchsten erzielten Score in der Matrix | |||
Score: Match: +3 | Mismatch: 0 | Gap: -1 | |||
[[File:Smith Waterman.jpg|600px|thumb|center]] | |||
== FASTA-Format == | |||
* FSTA ist ein Programm zur Suche von Sequenzen in Datenbanken also eine Alignment Software | |||
* das FASTA-Format ist ein allgemienes Format zur Speicherung von Sequenzdaten (Protein und DNA) in Textformat | |||
* das Format folg einem festen Aufbau: | |||
# Zeile: Sequenz ID, Zeile startet mit ">" | |||
# Zeile: (optional) Kommentare | |||
# Zeile: die Sequenz | |||
* besteht aus zwei bis drei Zeilen pro Sequenz | |||
* entstammt der FASTA Software, wird heutzutage aber als universelles Format in der Bioinformatik genutzt | |||
Latest revision as of 13:57, 26 September 2021
Sequenzalignment
Das optimale „Aneinander ausrichten“ von Sequenzen, sodass z.B. reads aus einer Sequenzierung an ein Referenzgenom ausgerichtet werden können.
Es gibt enorm viele Möglichkeiten eine Sequenz an eine andere zu alignen. Deshalb benötigt man spezielle Algorithmen, die die besten Möglichkeiten finden. Um zu bestimmen welches Alignment das beste ist, wird ein Bewertungsschema, der sogenannte alignment score, angewandt. So kann die Übereinstimmung zweier Sequenzen miteinander quantitativ (mit Zahlen) verglichen werden.
Es werden nicht alle möglichen Alignments ausprobiert und dann deren Scores verglichen, um herauszufinden welches Alignment das beste ist. Mit dieser "brute force" Methode ergeben sich bei kurzen Sequenzen von 100 Basen schon 1075 Alignmentmögichkeiten. Stattdessen werden Sequenzen mit Hilfe des "Dynamic Programming" alignt. Es folgt dem Prinzip von "divide and conquer", also die Zerlegung des Problems in viele Unterprobleme, die nacheinander gelöst werden.
Zudem ermöglicht das Sequenzalignment die Erkennung ähnlicher Domänen. Da oft sogar homologe Sequenzen, die von einer gemeinsamen Sequenz abstammen, durch indels, die im Laufe der Evolution aufgetreten sind, unterschiedliche Längen besitzen, ist es notwendig gaps Einzufügen um die Ähnlichkeit der beiden Sequenzen sichtbar zu machen (s. Smith-Waterman).
Anwenndungen finden Alignments im Sequenzvergeleich, z.B. bei phylogenetischen Untersuchungen.
Man unterscheidet zwei verschiedenen Arten beim Sequenzalignment:
Globales Alignment
- Alignment zwischen zwei Sequenzen, die ähnlich lang sind und bei denen starke Sequenzhomologien erwartet werden
- Vergleich von Gesamtsequenzen
- alle Symbole werden berücksichtigt
- zur Berechnung des optimalen Alignment Scores wird häufig der Needle-Wunsch Algorithmus benutzt
Beispiel:
| H | A | U | S | K | L | A | U | S | ||
| ↓ | ||||||||||
| H | - | A | U | S | - | H | A | U | S | |
| K | L | A | U | S | K | L | A | U | S | |
| Score 8 | Score 8 | |||||||||
Bewertungsschema:
S(a,b)={3 a=b match ;0 a≠b mismatch
| gaps: | S(a,-)=-1 Deletion |
| S(-,b)=-1 Insertion |
Lokales Alignment
- Alignment von Teilsequenzen
- Vergleich zweier sehr unterschiedlicher Sequenzen, die aber gleiche Motive besitzen
- z.B. die Suche einer Gensequenz im Genom
Beispiel:
| A | T | G | C | A | T | T | A | C |
| C | T | T | T | A |
Smith-Waterman Algorithmus
Dynamic programming: "divide and conquer", Aufteilen des Problems in Subprobleme
M0,0=0
M0,j=0
Mk,0=0
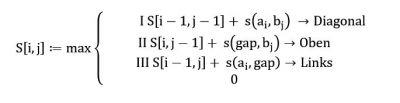
Das Alignment beginnt bei dem höchsten erzielten Score in der Matrix
Score: Match: +3 | Mismatch: 0 | Gap: -1
FASTA-Format
- FSTA ist ein Programm zur Suche von Sequenzen in Datenbanken also eine Alignment Software
- das FASTA-Format ist ein allgemienes Format zur Speicherung von Sequenzdaten (Protein und DNA) in Textformat
- das Format folg einem festen Aufbau:
- Zeile: Sequenz ID, Zeile startet mit ">"
- Zeile: (optional) Kommentare
- Zeile: die Sequenz
- besteht aus zwei bis drei Zeilen pro Sequenz
- entstammt der FASTA Software, wird heutzutage aber als universelles Format in der Bioinformatik genutzt