5 Transkriptom RNA Seq 2: Difference between revisions
From Bioinformatik Wiki
No edit summary |
No edit summary |
||
| (9 intermediate revisions by 3 users not shown) | |||
| Line 1: | Line 1: | ||
= RNAseq = | = RNAseq = | ||
RNA sequencing ist eine Methode zur Analyse von Transkriptomen. Hierbei wird die Sequenz aller in der Probe vorhandenen mRNA Moleküle durch Next-Generation Sequencing (NGS) ermittelt. | |||
<br> | <br> | ||
== 1. Isolierung von mRNA == | == 1. Isolierung von mRNA == | ||
* nur 1-2% der totalen RNA ist mRNA | * nur 1-2% der totalen RNA ist mRNA | ||
* 90% rRNA | * 90% rRNA | ||
** poly(A) Anreicherung | * weil wir nur die mRNA isolieren wollen, müssen wir diese von der restlichen RNA trennen | ||
** Abbau von rRNA | * das ist entweder durch die sogennante poly(A) Anreicherung möglich, dabeimacht man sich die besondere Eigenschaft der mRNA zu nutze | ||
* diese besitzt im Gegensatz zu anderen RNA Spezies einen polyA-Schwanz, immobilisiert man auf der Matrix einer Chromatofraphiesäule polyT-Seqeunezen, kann die mRNA komplementär an diese binden und kann so hufgereinigt werden | |||
* die zweite Möglichkeit ist der spezifische Abbau von rRNA | |||
* diese Vorgehensweise gewinnt vor allem bei Prokaryotischer mRNA an Bedeutung, bei der es keinen polyA-Schwanz gibt | |||
<br> | <br> | ||
<br> | <br> | ||
| Line 12: | Line 16: | ||
*Information über kodierenden Strang kann durch 2-stufige cDNA Synthese und dUTP Nukleotiden konserviert werden. | *Information über kodierenden Strang kann durch 2-stufige cDNA Synthese und dUTP Nukleotiden konserviert werden. | ||
<br> | <br> | ||
[[File:RNA-Seq.png|400px|center|thumb]] | |||
| | |||
* Abbau des non-coding Stranges durch durch Uracil-DNA-Glykosylase | |||
* baut spezifisch den non-coding Strang ab, weil dieser als einziger Uracil besitzt | |||
* d.h. alle zum cDNA-Originalstrang komplementären Stränge werden abgebaut | |||
* durch die Adapter bleibt die Topologie erhalten | |||
* es folgt die Amplifikation mittels PCR | |||
== 3. Sequenzierung/ Next generation Sequencing == | == 3. Sequenzierung/ Next generation Sequencing == | ||
* Sequenziertiefe bestimmt die Empfindlichkeit und die Genauigkeit | |||
* 5 Millionen reads ausreichend für mittel-hoch exprimierte Gene | |||
* 100 Millionen reads für schwach exprimierte Gene | |||
* wichtig: Anzahl an Replikaten bestimmt statistische Sensitivität für Unterschiede | |||
<big> | <big> | ||
| Line 59: | Line 47: | ||
|} | |} | ||
</big> | </big> | ||
== 4. | * die Tabelle stellt die Wahrscheinlichkeit dar, einen bestimmten fold change bei einer bestimmten Anzahl an Repliken zu delektieren bzw. statistisch signifikant zu finden | ||
== 4. Datenanalyse == | |||
Präsprozessierung der Rohdaten: | |||
<br> | <br> | ||
⇒ Filtern von Basen mit geringer | ⇒Filtern von Basen mit geringer Sequenzierqualität | ||
<br> | <br> | ||
⇒ Trimmen von Adaptersequenzen und PCR-Primersequenzen | ⇒ Trimmen von Adaptersequenzen und PCR-Primersequenzen | ||
<br> | <br> | ||
⇒ Programme: FASTQC, NGSQC, Trimmomatic | |||
Programme: FASTQC, NGSQC, Trimmomatic | |||
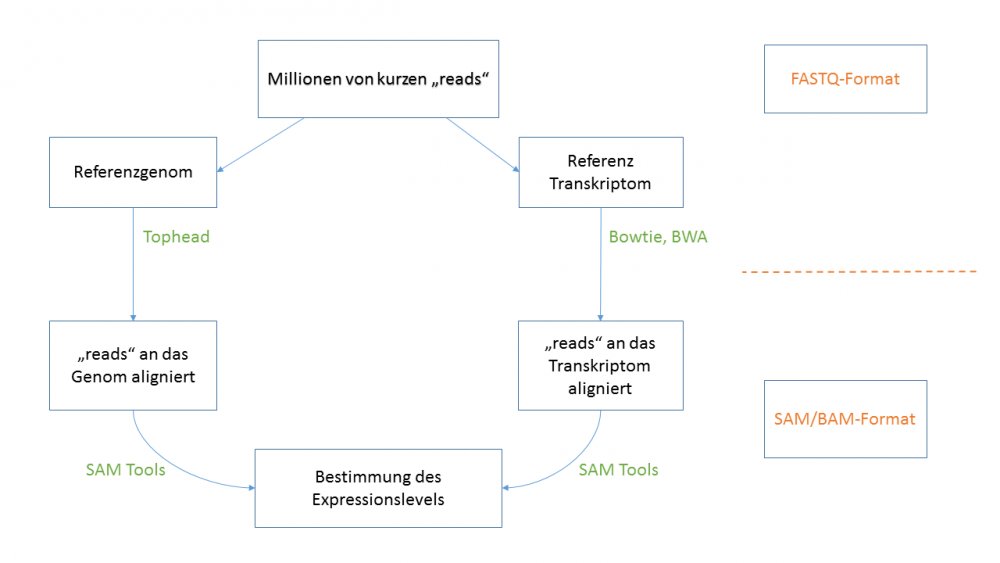
== 5. Read Alignment == | == 5. Read Alignment == | ||
[[File:Read-alignment.png| | [[File:Read-alignment.png|center|1000px]] | ||
=== SAM und BAM Format === | |||
* Enthält Informationen über die Sequenz-reads nach dem Alignment an ein Referenzgenom. | |||
* SAM= Sequence Alignment/Map | |||
* BAM= Binary Alignment/Map | |||
* SAM ist ein textbasiertes Dateiformat und BAM ein binäres Dateiformat. | |||
== Sequenziertiefe == | |||
Die Sequenziertiefe gibt an, wie oft ein bestimmter Abschnitt eines Genoms sequenziert wurde und bestimmt damit die Empfindlichkeit und Genauigkeit einer Sequenzierung. | |||
<center><math> Sequenziertiefe= \frac{\text{mapped reads * Fragmentlänge}}{\text{effective Genomgröße}}</math></center> | |||
Latest revision as of 14:20, 30 September 2021
RNAseq
RNA sequencing ist eine Methode zur Analyse von Transkriptomen. Hierbei wird die Sequenz aller in der Probe vorhandenen mRNA Moleküle durch Next-Generation Sequencing (NGS) ermittelt.
1. Isolierung von mRNA
- nur 1-2% der totalen RNA ist mRNA
- 90% rRNA
- weil wir nur die mRNA isolieren wollen, müssen wir diese von der restlichen RNA trennen
- das ist entweder durch die sogennante poly(A) Anreicherung möglich, dabeimacht man sich die besondere Eigenschaft der mRNA zu nutze
- diese besitzt im Gegensatz zu anderen RNA Spezies einen polyA-Schwanz, immobilisiert man auf der Matrix einer Chromatofraphiesäule polyT-Seqeunezen, kann die mRNA komplementär an diese binden und kann so hufgereinigt werden
- die zweite Möglichkeit ist der spezifische Abbau von rRNA
- diese Vorgehensweise gewinnt vor allem bei Prokaryotischer mRNA an Bedeutung, bei der es keinen polyA-Schwanz gibt
2. cDNA Synthese und Library Präparation
- Information über kodierenden Strang kann durch 2-stufige cDNA Synthese und dUTP Nukleotiden konserviert werden.
- Abbau des non-coding Stranges durch durch Uracil-DNA-Glykosylase
- baut spezifisch den non-coding Strang ab, weil dieser als einziger Uracil besitzt
- d.h. alle zum cDNA-Originalstrang komplementären Stränge werden abgebaut
- durch die Adapter bleibt die Topologie erhalten
- es folgt die Amplifikation mittels PCR
3. Sequenzierung/ Next generation Sequencing
- Sequenziertiefe bestimmt die Empfindlichkeit und die Genauigkeit
- 5 Millionen reads ausreichend für mittel-hoch exprimierte Gene
- 100 Millionen reads für schwach exprimierte Gene
- wichtig: Anzahl an Replikaten bestimmt statistische Sensitivität für Unterschiede
| Replikate pro Gruppe | |||
| fold change | 3 | 5 | 10 |
| 1,25 | 17% | 25% | 44% |
| 1,5 | 43% | 64% | 91% |
| 2 | 87% | 98% | 100% |
- die Tabelle stellt die Wahrscheinlichkeit dar, einen bestimmten fold change bei einer bestimmten Anzahl an Repliken zu delektieren bzw. statistisch signifikant zu finden
4. Datenanalyse
Präsprozessierung der Rohdaten:
⇒Filtern von Basen mit geringer Sequenzierqualität
⇒ Trimmen von Adaptersequenzen und PCR-Primersequenzen
⇒ Programme: FASTQC, NGSQC, Trimmomatic
5. Read Alignment
SAM und BAM Format
- Enthält Informationen über die Sequenz-reads nach dem Alignment an ein Referenzgenom.
- SAM= Sequence Alignment/Map
- BAM= Binary Alignment/Map
- SAM ist ein textbasiertes Dateiformat und BAM ein binäres Dateiformat.
Sequenziertiefe
Die Sequenziertiefe gibt an, wie oft ein bestimmter Abschnitt eines Genoms sequenziert wurde und bestimmt damit die Empfindlichkeit und Genauigkeit einer Sequenzierung.