6 Normalisierungen: Difference between revisions
No edit summary |
(Rest wird genauso formatiert, damit es gleichmässig aussieht) |
||
| (69 intermediate revisions by 5 users not shown) | |||
| Line 1: | Line 1: | ||
=Normalisierungen= | =Normalisierungen= | ||
Normalisierungen der RNASeq-Daten sind notwendig, um diese | Unter Normalisierung versteht man in der Biologie die Verringerung von Verzerrungen innerhalb eines Datensatzes. Systematische Verzerrungen sind z.B. verschiedene Durchführungsrunden, unterschiedliche Bedingungen, Laborausstattung, Experimentatoren ect. | ||
Auch die heterologe Geneigenschaften können zu unterschiedlichen Ergebnissen bei eigentlich gleicher Genexpression führen, wie z.B. Genlänge. | |||
Normalisierungen der RNASeq-Daten sind deshalb notwendig, um diese vergleichen. zu können und die tatsächliche Genexpression quantifizieren zu können. | |||
==RPKM== | ==RPKM== | ||
RPKM steht für 'Reads per kilobase of transcript per Million mapped reads' | RPKM steht für 'Reads per kilobase of transcript per Million mapped reads' und ist eine Normalisierungsmethode für Expressionsstärken. <br> | ||
{| class="wikitable" | |||
|- | |||
! Formel !! Parameter | |||
|- | |||
| <math> RPKM = \frac{ c_\text{i}}{L_\text{i} \cdot N} *10^6</math> || c<sub>i</sub> = Anzahl an ausrichtbaren reads für ein Transkript 'i' | |||
L<sub>i</sub> = Länge des Transkripts/Gens 'i' in kbp <br> | |||
N = Gesamtanzahl an ausrichtbaren reads in Millionen | |||
|} | |||
<math>\frac{ c_\text{i}}{L_\text{i}}</math> → Normalisierung der Genlänge <br> | |||
<math> L_\text{i} \cdot N </math> → Normalisierung der Sequenziertiefe | |||
Je höher der RPKM-Wert ist, desto höher ist die Genexpression. <br> | |||
RPKM-Werte können gut zum Vergleich der Genexpressionen innerhalb einer Probe genutzt werden, jedoch sind sie nicht dafür geeignet mehrere Experimente bzw. Proben miteinander zu vergleichen. | |||
'''Beispiel:''' | |||
[[File:Beispiel Probe.png|Beispiel Probe]] <br><br> | |||
<math> N=6*10^6 reads </math> <br> | |||
* <math> RPKM (A) = \frac{12}{0,6 kbp \cdot 6} * 10^6 = 3,33 \cdot 10^{6} </math> <br> | |||
* <math> RPKM (B) = \frac{24}{1,1 kbp \cdot 6} * 10^6 = 3,64 \cdot 10^{6} </math> <br> | |||
* <math> RPKM (C) = \frac{11}{1,4 kbp \cdot 6} * 10^6= 1,31 \cdot 10^{6} </math> <br><br> | |||
Durch die Normalisierung der Expressionsstärken können diese nun miteinander vergleichen werden.In diesem Beispiel wird Gen B am stärksten exprimiert und Gen C am schwächsten. | |||
==TPM== | ==TPM== | ||
TPM steht für 'Transcripts per Million' | TPM steht für 'Transcripts per Million' und ist eine Normalisierungsmethode für Expressionsstärken. <br> | ||
{| class="wikitable" | |||
|- | |||
! Formel !! Parameter | |||
|- | |||
|<math> TPM = \frac{ c_\text{i}}{L_\text{i}} \cdot \dfrac {1}{\sum_\text{n} \dfrac {c_\text{n}}{L_\text{n}}} \cdot 10^6 </math> <br> || c<sub>i</sub> = Anzahl an ausrichtbaren reads für ein Transkript 'i'<br> | |||
L<sub>i</sub> = Länge des Transkripts/Gens 'i' in bp <br> | |||
N = Gesamtanzahl an ausrichtbaren reads in Millionen | |||
|} | |||
Wenn 1 Millionen Transkripte sequenziert würden, wäre TPM<sub>i</sub> die Anzahl an Transkripten des Types 'i'. <br> | |||
'''Beispiel:''' <br> | |||
[[File:Beispiel Probe.png|Beispiel Probe]] <br><br> | |||
*<math> TPM (A) = \frac{ 12}{600} \cdot \dfrac {1}{ \dfrac {12}{600} + \dfrac {24}{1100} + \dfrac {11}{1400}} \cdot 10^6 = 402614 </math> <br> | |||
*<math> TPM (B) = \frac{ 24}{1100} \cdot \dfrac {1}{ \dfrac {12}{600} + \dfrac {24}{1100} + \dfrac {11}{1400}} \cdot 10^6 = 439216 </math> <br> | |||
*<math> TPM (C) = \frac{ 11}{1400} \cdot \dfrac {1}{ \dfrac {12}{600} + \dfrac {24}{1100} + \dfrac {11}{1400}} \cdot 10^6 = 158170 </math> <br> | |||
Nach der Normalisierung ist ein Vergleich der Genexpressionen möglich. Das Ergebnis gibt an, wie viele Transkripte der Gene entstehen würden, wenn 1 Millionen Transkripte sequenziert werden würden. So würde man z.B. für Gen B 429216 Transkripte erhalten und für Gen C 158170 Transkripte, somit wird Gen B stärker exprimiert, als Gen C. Die Summe der reads ergibt 1 Million. | |||
'''Probleme von RPKM und TPM:''' | |||
Die beiden Methoden sind nur gut geeignet, um Expressionswerte bzw. Expressionslevel innerhalb einer Probe zu vergleichen.Von sehr heterologen Expressionsprofilen zwischen zwei Proben können identische Genexpressionen fälschlicherweise als unterschiedlich erkannt werden. | |||
Lösung: TMM als Normalisierungsmethode zwischen Proben | |||
==TMM== | ==TMM== | ||
TMM steht für 'Trimmed mean of M-values normalization method' | TMM steht für 'Trimmed mean of M-values normalization method'.<br> | ||
*Gesamtexpressionsstärke pro Probe wird bestimmt, um einzelne Genexpressionen zu bestimmen. | |||
*Das Expressionsverhältnis zwischen zwei Bedingungen für ein Gen 'g' wird bestimmt. | |||
*Genexpressionen von zwei Proben mit unterschiedlichen Bedingungen (z.B. krank und gesund) können miteinander verglichen werden.<br> | |||
'''1.''' Bestimmung des '''Gewichtungsfaktors''' für das Expressionsverhältnis für Gen 'g' zwischen Probe 'k' ' und 'k'. Dabei werden kleinere Werte verstärkt und große abgeschwächt. <br> | |||
{| class="wikitable" | |||
|- | |||
! Formel !! Parameter | |||
|- | |||
| <math> W_\text{gk}^\text{k`} = \frac{ N_\text{k} - Y_\text{gk}}{N_\text{k} \cdot Y_\text{gk}} + \frac{ N_\text{k`} - Y_\text{gk`}}{N_\text{k`} \cdot Y_\text{gk`}} </math> || Y<sub>gk</sub> = Beobachtete Anzahl an reads für Gen 'g' und Probe 'k' <br> | |||
N<sub>k</sub> = Gesamtanzahl an reads für Probe 'k' <br> | |||
k' = 2. Probe (Referenz-Bedingung) | |||
|} | |||
'''2. Mittelwert aller Expressionsverhältnisse''' wird berechnet. <br> | |||
{| class="wikitable" | |||
|- | |||
! Formel !! Parameter | |||
|- | |||
| <math> M_\text{gk}^\text{k`} = log_2 \dfrac { \dfrac {Y_\text{gk}}{N_\text{k}}}{ \dfrac {Y_\text{gk`}}{N_\text{k`}}} </math> || Y<sub>gk</sub> = Beobachtete Anzahl an reads für Gen 'g' und Probe 'k' <br> | |||
N<sub>k</sub> = Gesamtanzahl an reads für Probe 'k' <br> | |||
k' = 2. Probe (Referenz-Bedingung) | |||
|} | |||
'''3. Gewichtete Mittelwerte aller Expressionsverhältnisse''' können berechnet werden. Einsetzen der in 1. und 2. ermittelten Werte.<br> | |||
<div align="center"> <math> TMM = 2^{log_2 (TMM_\text{k}^\text{k`})} </math></div> <br> | |||
'''Gesamt:''' | |||
{| class="wikitable" | |||
|- | |||
! Formel !! Parameter | |||
|- | |||
| <math> log_2 (TMM_\text{k}^\text{k`}) = \frac{ \underset {g \in G}{\sum} W_\text{gk}^\text{k´} \cdot M_\text{gk}^\text{k´}} { \sum_{g \in G} W_\text{gk}^\text{k´}} </math> || <math> M_\text{gk}^\text{k`} = </math> Mittelwerte aller Expressionsverhältnisse <br> | |||
<math> W_\text{gk}^\text{k`} = </math> Gewichtungsfaktor der Expressionsverhältnisse <br> | |||
G = Set aller Gene | |||
|} | |||
'''Bedingungen für dieses Verfahren:''' <br> | |||
* Y<sub>gk</sub> , Y<sub>gk'</sub> > 0 | |||
* Die oberen und unteren 30 % der <math> M_\text{gk}^\text{k`} </math> Werte werden nicht berücksichtigt | |||
* Die 5 % am höchsten und niedrigsten exprimierten Gene werden nicht berücksichtigt | |||
== Beispielrechnung == | |||
Gen Zustand 1[bp] Zustand 2[bp] | |||
A 200 600 | |||
B 600 0 | |||
C 800 960 | |||
D 400 480 | |||
Summe 2000 1680 | |||
Folgende Dinge gelten nun: | |||
* TMM braucht immer einen Referenzwert (hier: Zustand 1); Wenn man mehrere Proben hätte, könnte man diese alle an Zustand 1 messen. | |||
* Zuerst normalisiert man die Counts innerhalb der Probe | |||
Ya/N Yb/N<br> | |||
A <math>\frac{200}{2000}=0,1</math> <math>\frac{240}{1680}=0,143</math><br> | |||
B <math>\frac{600}{2000}=0,3</math> <math>\frac{0}{1680}=0</math><br> | |||
C <math>\frac{800}{2000}=0,4</math> <math>\frac{960}{1680}=0,571</math><br> | |||
D <math>\frac{400}{2000}=0,2</math> <math>\frac{2480}{1680}=0,286</math> | |||
* Mittelwert der Expressionsverhältnisse | |||
Ma/b | |||
A <math>log_{2} \frac{0,143}{0,1}=0,516</math><br> | |||
B <math>log_{2} \frac{0}{0,3}= -Inf </math> <br> | |||
C <math>log_{2} \frac{0,571}{0,4}=0,513</math><br> | |||
D <math>log_{2} \frac{0,286}{0,2}=0,516</math><br> | |||
*'''kleiner Praxistipp von eurem Admin''': <math>log_{2}</math> ist der Logarithmus von 2, Es ist nicht der natürliche log, es ist nicht ln, es ist nichts mit eulerscher Zahl oder was eure Taschenrechner sonst noch so hergeben. Also sucht eine Funktion, in der ihr die Basis des log eintragen könnt und gebt dort eine 2 ein. | |||
*Nun kommt der Gewichtungsfaktor ins Spiel | |||
Wa/b | |||
A <math> \frac{1680-240}{1680*240} + \frac{2000-200}{2000*200}=0,081</math><br> | |||
B <math> \frac{1680-0}{1680*0} + \frac{2000-600}{2000*600}= -Inf </math><br> | |||
C <math> \frac{1680-960}{1680*960} + \frac{2000-800}{2000*800}=0,0012</math><br> | |||
D <math> \frac{1680-480}{1680*480} + \frac{2000-400}{2000*400}=0,0035</math><br> | |||
'''Summe der Gewichtung 0,0128''' | |||
*Nun werden die Mittelwerte der Expressionsverhältnisse gewichtet (durch den Gewichtungsfaktor) | |||
A 0,516 * 0,081 = 0,0042 | |||
B - | |||
C 0,513 * 0,0012 = 0,0006 | |||
D 0,516 * 0,0018 = 0,0018 | |||
Summe 0,0066 | |||
Nun wird TMM berechnet:<br> | |||
<math>\frac{\text{Summe der gewichteten Mittelwerte der Expressionsverhältnisse}}{\text{Summe Gewichtungsfaktoren}}=log_{2}(TMM)</math> | |||
<math>\frac{0,0066}{0,0128}=0,51=log_{2}(TMM)</math><br> | |||
* Jetzt noch die Formel umsetzen: | |||
<math>log_{2}(TMM)= x</math> | |||
<math>2^{x} = TMM </math> | |||
<math>2^{0,51} = 1,4296 </math> | |||
Latest revision as of 14:58, 11 June 2024
Normalisierungen
Unter Normalisierung versteht man in der Biologie die Verringerung von Verzerrungen innerhalb eines Datensatzes. Systematische Verzerrungen sind z.B. verschiedene Durchführungsrunden, unterschiedliche Bedingungen, Laborausstattung, Experimentatoren ect. Auch die heterologe Geneigenschaften können zu unterschiedlichen Ergebnissen bei eigentlich gleicher Genexpression führen, wie z.B. Genlänge. Normalisierungen der RNASeq-Daten sind deshalb notwendig, um diese vergleichen. zu können und die tatsächliche Genexpression quantifizieren zu können.
RPKM
RPKM steht für 'Reads per kilobase of transcript per Million mapped reads' und ist eine Normalisierungsmethode für Expressionsstärken.
| Formel | Parameter |
|---|---|
| [math]\displaystyle{ RPKM = \frac{ c_\text{i}}{L_\text{i} \cdot N} *10^6 }[/math] | ci = Anzahl an ausrichtbaren reads für ein Transkript 'i'
Li = Länge des Transkripts/Gens 'i' in kbp |
[math]\displaystyle{ \frac{ c_\text{i}}{L_\text{i}} }[/math] → Normalisierung der Genlänge
[math]\displaystyle{ L_\text{i} \cdot N }[/math] → Normalisierung der Sequenziertiefe
Je höher der RPKM-Wert ist, desto höher ist die Genexpression.
RPKM-Werte können gut zum Vergleich der Genexpressionen innerhalb einer Probe genutzt werden, jedoch sind sie nicht dafür geeignet mehrere Experimente bzw. Proben miteinander zu vergleichen.
Beispiel:
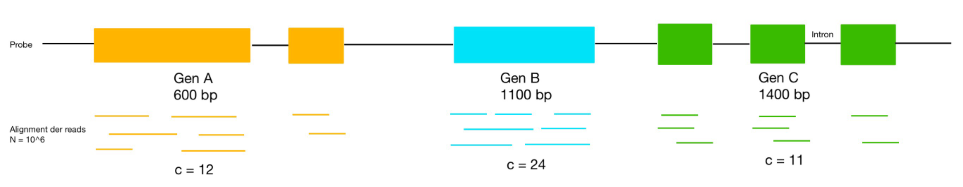
[math]\displaystyle{ N=6*10^6 reads }[/math]
- [math]\displaystyle{ RPKM (A) = \frac{12}{0,6 kbp \cdot 6} * 10^6 = 3,33 \cdot 10^{6} }[/math]
- [math]\displaystyle{ RPKM (B) = \frac{24}{1,1 kbp \cdot 6} * 10^6 = 3,64 \cdot 10^{6} }[/math]
- [math]\displaystyle{ RPKM (C) = \frac{11}{1,4 kbp \cdot 6} * 10^6= 1,31 \cdot 10^{6} }[/math]
Durch die Normalisierung der Expressionsstärken können diese nun miteinander vergleichen werden.In diesem Beispiel wird Gen B am stärksten exprimiert und Gen C am schwächsten.
TPM
TPM steht für 'Transcripts per Million' und ist eine Normalisierungsmethode für Expressionsstärken.
| Formel | Parameter |
|---|---|
| [math]\displaystyle{ TPM = \frac{ c_\text{i}}{L_\text{i}} \cdot \dfrac {1}{\sum_\text{n} \dfrac {c_\text{n}}{L_\text{n}}} \cdot 10^6 }[/math] |
ci = Anzahl an ausrichtbaren reads für ein Transkript 'i' Li = Länge des Transkripts/Gens 'i' in bp |
Wenn 1 Millionen Transkripte sequenziert würden, wäre TPMi die Anzahl an Transkripten des Types 'i'.
- [math]\displaystyle{ TPM (A) = \frac{ 12}{600} \cdot \dfrac {1}{ \dfrac {12}{600} + \dfrac {24}{1100} + \dfrac {11}{1400}} \cdot 10^6 = 402614 }[/math]
- [math]\displaystyle{ TPM (B) = \frac{ 24}{1100} \cdot \dfrac {1}{ \dfrac {12}{600} + \dfrac {24}{1100} + \dfrac {11}{1400}} \cdot 10^6 = 439216 }[/math]
- [math]\displaystyle{ TPM (C) = \frac{ 11}{1400} \cdot \dfrac {1}{ \dfrac {12}{600} + \dfrac {24}{1100} + \dfrac {11}{1400}} \cdot 10^6 = 158170 }[/math]
Nach der Normalisierung ist ein Vergleich der Genexpressionen möglich. Das Ergebnis gibt an, wie viele Transkripte der Gene entstehen würden, wenn 1 Millionen Transkripte sequenziert werden würden. So würde man z.B. für Gen B 429216 Transkripte erhalten und für Gen C 158170 Transkripte, somit wird Gen B stärker exprimiert, als Gen C. Die Summe der reads ergibt 1 Million.
Probleme von RPKM und TPM:
Die beiden Methoden sind nur gut geeignet, um Expressionswerte bzw. Expressionslevel innerhalb einer Probe zu vergleichen.Von sehr heterologen Expressionsprofilen zwischen zwei Proben können identische Genexpressionen fälschlicherweise als unterschiedlich erkannt werden.
Lösung: TMM als Normalisierungsmethode zwischen Proben
TMM
TMM steht für 'Trimmed mean of M-values normalization method'.
- Gesamtexpressionsstärke pro Probe wird bestimmt, um einzelne Genexpressionen zu bestimmen.
- Das Expressionsverhältnis zwischen zwei Bedingungen für ein Gen 'g' wird bestimmt.
- Genexpressionen von zwei Proben mit unterschiedlichen Bedingungen (z.B. krank und gesund) können miteinander verglichen werden.
1. Bestimmung des Gewichtungsfaktors für das Expressionsverhältnis für Gen 'g' zwischen Probe 'k' ' und 'k'. Dabei werden kleinere Werte verstärkt und große abgeschwächt.
| Formel | Parameter |
|---|---|
| [math]\displaystyle{ W_\text{gk}^\text{k`} = \frac{ N_\text{k} - Y_\text{gk}}{N_\text{k} \cdot Y_\text{gk}} + \frac{ N_\text{k`} - Y_\text{gk`}}{N_\text{k`} \cdot Y_\text{gk`}} }[/math] | Ygk = Beobachtete Anzahl an reads für Gen 'g' und Probe 'k' Nk = Gesamtanzahl an reads für Probe 'k' |
2. Mittelwert aller Expressionsverhältnisse wird berechnet.
| Formel | Parameter |
|---|---|
| [math]\displaystyle{ M_\text{gk}^\text{k`} = log_2 \dfrac { \dfrac {Y_\text{gk}}{N_\text{k}}}{ \dfrac {Y_\text{gk`}}{N_\text{k`}}} }[/math] | Ygk = Beobachtete Anzahl an reads für Gen 'g' und Probe 'k' Nk = Gesamtanzahl an reads für Probe 'k' |
3. Gewichtete Mittelwerte aller Expressionsverhältnisse können berechnet werden. Einsetzen der in 1. und 2. ermittelten Werte.
Gesamt:
| Formel | Parameter |
|---|---|
| [math]\displaystyle{ log_2 (TMM_\text{k}^\text{k`}) = \frac{ \underset {g \in G}{\sum} W_\text{gk}^\text{k´} \cdot M_\text{gk}^\text{k´}} { \sum_{g \in G} W_\text{gk}^\text{k´}} }[/math] | [math]\displaystyle{ M_\text{gk}^\text{k`} = }[/math] Mittelwerte aller Expressionsverhältnisse [math]\displaystyle{ W_\text{gk}^\text{k`} = }[/math] Gewichtungsfaktor der Expressionsverhältnisse |
Bedingungen für dieses Verfahren:
- Ygk , Ygk' > 0
- Die oberen und unteren 30 % der [math]\displaystyle{ M_\text{gk}^\text{k`} }[/math] Werte werden nicht berücksichtigt
- Die 5 % am höchsten und niedrigsten exprimierten Gene werden nicht berücksichtigt
Beispielrechnung
Gen Zustand 1[bp] Zustand 2[bp] A 200 600 B 600 0 C 800 960 D 400 480 Summe 2000 1680
Folgende Dinge gelten nun: * TMM braucht immer einen Referenzwert (hier: Zustand 1); Wenn man mehrere Proben hätte, könnte man diese alle an Zustand 1 messen.
- Zuerst normalisiert man die Counts innerhalb der Probe
Ya/N Yb/N
A [math]\displaystyle{ \frac{200}{2000}=0,1 }[/math] [math]\displaystyle{ \frac{240}{1680}=0,143 }[/math]
B [math]\displaystyle{ \frac{600}{2000}=0,3 }[/math] [math]\displaystyle{ \frac{0}{1680}=0 }[/math]
C [math]\displaystyle{ \frac{800}{2000}=0,4 }[/math] [math]\displaystyle{ \frac{960}{1680}=0,571 }[/math]
D [math]\displaystyle{ \frac{400}{2000}=0,2 }[/math] [math]\displaystyle{ \frac{2480}{1680}=0,286 }[/math]
- Mittelwert der Expressionsverhältnisse
Ma/b
A [math]\displaystyle{ log_{2} \frac{0,143}{0,1}=0,516 }[/math]
B [math]\displaystyle{ log_{2} \frac{0}{0,3}= -Inf }[/math]
C [math]\displaystyle{ log_{2} \frac{0,571}{0,4}=0,513 }[/math]
D [math]\displaystyle{ log_{2} \frac{0,286}{0,2}=0,516 }[/math]
- kleiner Praxistipp von eurem Admin: [math]\displaystyle{ log_{2} }[/math] ist der Logarithmus von 2, Es ist nicht der natürliche log, es ist nicht ln, es ist nichts mit eulerscher Zahl oder was eure Taschenrechner sonst noch so hergeben. Also sucht eine Funktion, in der ihr die Basis des log eintragen könnt und gebt dort eine 2 ein.
- Nun kommt der Gewichtungsfaktor ins Spiel
Wa/b
A [math]\displaystyle{ \frac{1680-240}{1680*240} + \frac{2000-200}{2000*200}=0,081 }[/math]
B [math]\displaystyle{ \frac{1680-0}{1680*0} + \frac{2000-600}{2000*600}= -Inf }[/math]
C [math]\displaystyle{ \frac{1680-960}{1680*960} + \frac{2000-800}{2000*800}=0,0012 }[/math]
D [math]\displaystyle{ \frac{1680-480}{1680*480} + \frac{2000-400}{2000*400}=0,0035 }[/math]
Summe der Gewichtung 0,0128
- Nun werden die Mittelwerte der Expressionsverhältnisse gewichtet (durch den Gewichtungsfaktor)
A 0,516 * 0,081 = 0,0042 B - C 0,513 * 0,0012 = 0,0006 D 0,516 * 0,0018 = 0,0018 Summe 0,0066
Nun wird TMM berechnet:
[math]\displaystyle{ \frac{\text{Summe der gewichteten Mittelwerte der Expressionsverhältnisse}}{\text{Summe Gewichtungsfaktoren}}=log_{2}(TMM) }[/math]
[math]\displaystyle{ \frac{0,0066}{0,0128}=0,51=log_{2}(TMM) }[/math]
- Jetzt noch die Formel umsetzen:
[math]\displaystyle{ log_{2}(TMM)= x }[/math] [math]\displaystyle{ 2^{x} = TMM }[/math] [math]\displaystyle{ 2^{0,51} = 1,4296 }[/math]