2 Transkriptom RNA Seq 1: Difference between revisions
| Line 23: | Line 23: | ||
== Sequenzierung == | == Sequenzierung == | ||
Für die quantitativen und qualitativen Bestimmungen von Sequenzen bei der Transkriptomanalyse müssen die cDNA-Sequenzen sequenziert werden. | Für die quantitativen und qualitativen Bestimmungen von Sequenzen bei der Transkriptomanalyse durch die RNA-Sequenzierung müssen, wie der Name schon verrät, die cDNA-Sequenzen sequenziert werden. | ||
=== First Generation === | === First Generation === | ||
Revision as of 11:49, 21 September 2021
Biologische Fragestellung
Das NAT8L Gen scheint im Metabolismus von Krebszellen eine Rolle zu spielen. Unser Projekt befasst sich mit Lungenkrebs. Daraus folg folgende Frage: Ist NAT8L in Lungenkrebszellen erhöht exprimiert ?
Die Frage impliziert weitere Überlegungen z.B. ob NAT8L essentiell für das Wachstum von Krebszellen ist und ob eine Inhibierung der NAT8L Expression die Tumorbildung hemmen würde.
Um die Frage zu beantworten sind folgende drei Schritte notwendig:
Schritt 1:
- Abfragen von Datenbanken (vielleicht wurden bereits ähnliche Experimente durchgeführt)
Schritt 2:
- aus Datenbanken haben wir herausgefunden, dass NAT8L in Lungenkrebszellen erhöht expliziert wird
Schritt 3:
- in einem Experiment soll versucht werden die Genexpression durch RNAi (RNA interference) zu silencen
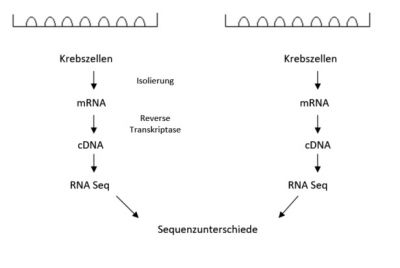
- aus zwei Tumorzellkulturen wird die mRNA extrahiert
- mit Hilfe der reversen Transkriptase wird cDNA transkribiert
- cDNA wird sequenziert
- Sequenzunterschiede können analysiert werden
Dem Experiment liegt die Transkriptomanalyse zugrunde, welche eine quantitative und qualitative Bestimmung aller Transkriptionsaktivitäten ermöglicht. D.h. die Erfassung aller mRNA-Moleküle, wodurch auf die Aktivität bestimmter Gene unter bestimmten Bedingungen zurückschließen lässt. Zum Ablauf der Transkriptomanalyse durch RNA-Sequenzierung vergleiche 5.Transkriptom RNA Seq 2
Sequenzierung
Für die quantitativen und qualitativen Bestimmungen von Sequenzen bei der Transkriptomanalyse durch die RNA-Sequenzierung müssen, wie der Name schon verrät, die cDNA-Sequenzen sequenziert werden.
First Generation
Sanger Methode
400px][https://de.wikipedia.org/wiki/DNA-Sequenzierung#Didesoxymethode_nach_Sanger
Next Generation Sequencing (NGS)
Illumina Sequencing (2nd Generation Sequencing)

Ergebnis der Illumina Sequenzierung:
- Länge der reads 50-600bp
- Fehlerrate ca. 0,1%
- humanes Genom kann 30x am Tag sequenziert werden
- Daten werden in FASTQ Format geliefert
FASTQ
Die NGS-Sequenzierergebnisse werden in diesem textbasierten-Format geliefert, dass zur Speicherung von DNA/RNA-Sequenzen dient.
Eine FASTQ-Datei ist folgendermaßen aufgebaut:
@ Identifier # Sequenz identifier GATCTT # Sequenz + # optionale Beschreibung !'CC'*+*!? # Qualität jedes Nukleotids (Zahlenwert repräsentiert durch ASCII Tabelle)
Problem
Viele kurze reads, die in einen Zusammenhang gebracht werden müssen! Um Lücken in der Sequenz zu vermeiden, muss die Sequenziertiefe angepasst werden.
Sequenzabdeckung
G: Länge der Genomsequenz
N: Anzahl der reads
L: durchschnittliche Länge der reads
C: Coverage (Abdeckung)
[math]\displaystyle{ C= \frac{N*L}{G} }[/math]
Lander-Waterman-Modell
Mathematisches Modell zur Errechnung, durch Sequenzierung, nicht abgedeckter Basenpaare.
P[nicht abgedecktes Bp] = e-c
Beispiele:
C=10 → 1 Gap in 22000 Bp
C=22 → 1 Gap in 3,6*109 Bp
C=30 → So tief, dass quasi alles überdeckt wird
Assemblierung der reads
Aus der RNA-Seq erhaltene Fragmente sequenzierter RNA. Diese werden anschließend dem Referenzgenom (Template) zugeordnet werden.
| -A | -C | -C | -T | -G | -A | -C | T | -A | -G | -C | -T | -G | -A | -T | -C | -A | -A | -G | -G | - | - | Template | |||
| -G | -A | -T | -C | -A | -A | ||||||||||||||||||||
| -A | -G | -C | -T | -G | -A | ||||||||||||||||||||
| -A | -C | G | -A | -G | -C | -T | - | - | Punktmutation | ||||||||||||||||
| -G | -A | -_ | -C | -A | -A | -G | -G | - | - | Deletion |
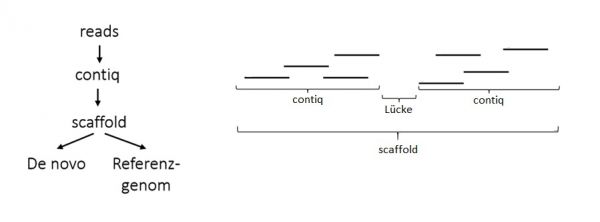
Damit die reads in Zusammenhang gebracht werden können müssen diese assembliert werden. Assembly ist ein Bioinformatisches Verfahren, bei dem überlappende reads zu Contiqs und anschließend zu Scaffolds zusammengesetzt werden. Wenn das Genom bereits bekannt ist können die Scaffolds an ein Referenzgenom aligned werden:
Contiq: Zusammenhängender Abschnitt einer genomischen Sequenz, die aus überlappenden reads gebildet wird. Zwischen ihnen befinden sich noch Lücken.
Scaffold: Bestehend aus Contiqs und Lücken (auch Supercontiqs genannt); definieren die Reihenfolge, Orientierung und Größe der Lücken zwischen den Contiqs.