9 Biomarker: Difference between revisions
No edit summary |
|||
| (9 intermediate revisions by 3 users not shown) | |||
| Line 6: | Line 6: | ||
[[File:VL8 1.png|mini|850px]] | [[File:VL8 1.png|mini|850px]] | ||
Wie in der Abbildung oben dargestellt, kann über Einzelwerte keine Aussage getroffen werden, ob der Patient krank oder gesund ist. <br> | Wie in der Abbildung oben dargestellt, kann über Einzelwerte keine Aussage getroffen werden, ob der Patient krank oder gesund ist. <br> | ||
→ deswegen bedarf es mehrerer Biomarker <br><br> | → deswegen bedarf es mehrerer Biomarker <br><br> | ||
'''Frage:''' <br> | '''Frage:''' <br> | ||
Wie kann für einen TPM-Wert vorhergesagt werden, ob es sich um 'Krebs' oder ' | Wie kann für einen TPM-Wert vorhergesagt werden, ob es sich um 'Krebs' oder 'Gesund' handelt?<br> | ||
==Logistische Funktion== | ==Logistische Funktion== | ||
<math> y = \beta_1 \cdot TPM_{HIF1\alpha} + \beta_0 </math> <br><br> | <math> y = \beta_1 \cdot TPM_{HIF1\alpha} + \beta_0 </math> <br><br> | ||
Damit wird die Wahrscheinlichkeit, dass jemand z.B. Krebs bekommt durch Gegenwahrscheinlichkeit bestimmt. <br> | |||
<math> logit_{(P)} = log \frac{P}{1-P} </math><br> | |||
Der Wertebereich der Logit Funktion ist ideal für lineare Regression | |||
<math> log \frac{P(x)}{1-P(x)} = \beta_1 \cdot TPM_{HIF1\alpha} + \beta_0 </math><br><br> | |||
[[File:LogFunktion.png|frameless|center]] | |||
==Biomarkersignatur== | ==Biomarkersignatur== | ||
Erweiterung für mehrere Gene: | Erweiterung für mehrere Gene: | ||
<math> P(x) = \frac{1}{e^{-((\beta)_1 \cdot x_1) + ((\beta)_2 \cdot x_1) + .... + (\beta)_n \cdot x_n) + \beta_0)}}</math> <br> | <math> P(x) = \frac{1}{e^{-((\beta)_1 \cdot x_1) + ((\beta)_2 \cdot x_1) + .... + (\beta)_n \cdot x_n) + \beta_0)}}</math> <br><br> | ||
'''Mashine learning''' <br> | '''Mashine learning''' <br> | ||
* Ein Modell muss zunächst von einem System erlent werden, dann kann das System eine Aussage treffen, ob z.B. 'krank' oder 'gesund'. <br> | * Ein Modell muss zunächst von einem System erlent werden, dann kann das System eine Aussage treffen, ob z.B. 'krank' oder 'gesund'. <br> | ||
* Parameter <math> \beta_1 </math> und <math> \beta_0 </math> können nicht berechnet werden. → Müssen mit Hilfe eines Algorithmus geschätzt werden. <br> <br> | * Parameter <math> \beta_1 </math> und <math> \beta_0 </math> können nicht berechnet werden. → Müssen mit Hilfe eines Algorithmus geschätzt werden. <br> <br> | ||
'''Maximum-Likelihood:''' <br> | '''Maximum-Likelihood:''' <br> | ||
* Schätzung zur Parameterbestimmung. <br> | * Schätzung zur Parameterbestimmung. <br> | ||
* ''Training'': Lerndaten sind bekannte Werte zur Aussage 'krank' oder 'gesund' <br> | * ''Training'': Lerndaten sind bekannte Werte zur Aussage 'krank' oder 'gesund' <br> | ||
→ Algorithmus bestimmt einen genauen Wert für <math> \beta_1</math>, um krank (1) und gesund (0) zu bestimmen. <br> | → Algorithmus bestimmt einen genauen Wert für <math> \beta_1</math>, um krank (1) und gesund (0) zu bestimmen. <br> | ||
→ Danach kann mit dem Modell bestimmt werden, ob es gut oder schlecht ist. <br> | → Danach kann mit dem Modell bestimmt werden, ob es gut oder schlecht ist. <br><br> | ||
'''Problem''': Das System versucht einen optimalen Wert zu finden <br> | |||
→ ''Overfitting'': Zu genaue Beschreibung des Modells.<br><br> | → ''Overfitting'': Zu genaue Beschreibung des Modells.<br><br> | ||
'''Frage:''' Wie gut ist die Qualität des Modells? <br> | '''Frage:''' Wie gut ist die Qualität des Modells? <br> | ||
==Receiver Operation Characteristic (ROC)== | ==Receiver Operation Characteristic (ROC)== | ||
Wird genutzt um zu prüfen, ob das Modell gut ist. <br> | Wird genutzt um zu prüfen, ob das Modell gut ist. <br> | ||
| Line 59: | Line 66: | ||
==Testen auf Spezifität== | ==Testen auf Spezifität== | ||
Möglichst viele 'Gesunde' sollen richtig bestimmt werden. <br> | Möglichst viele 'Gesunde' sollen richtig bestimmt werden. <br> | ||
<math> \text{Spezifität} = \frac{\text{TN}}{\text{ FP + TN}} </math> | <math> \text{Spezifität} = \frac{\text{TN}}{\text{ FP + TN}} </math> | ||
*Entweder hohe Sensitivität oder hohe Spezifität. | * Bei einem perfekten Modell gibt dieses keine falsch positiven Ergebnisse aus. <br> | ||
* Ausgabe 1 = Perfektes Modell, erkennt alle mit gesund. <br> | |||
* Ausgabe 0 = Schlechtes Modell, erkennt keine gesunden. <br> | |||
*Entweder hohe Sensitivität oder hohe Spezifität. Was besser ist kommt auf die Fragestellung an. <br> | |||
'''Treshold''': <br> | '''Treshold''': <br> | ||
Cutoff-Wert der als Grenzwert festgelegt wird nach dem beurteilt wird, ob der zu bewertende Wert 'krank', oder 'gesund' zugeordnet wird. <br> | Cutoff-Wert der als Grenzwert festgelegt wird nach dem beurteilt wird, ob der zu bewertende Wert 'krank', oder 'gesund' zugeordnet wird. <br> | ||
'''Beispiel zur Bestimmung des optimalen Treshold''' <br> | |||
gesund=0 und krank=1 | |||
{| class="wikitable" | {| class="wikitable" | ||
| || || colspan="6" style="text-align:center"| Treshold | |||
|- | |- | ||
! Zustand !! Ausgabe Modell !! 0 !! 0,25 !! 0,45 !! 0,55 !! 0,75 | ! Zustand !! Ausgabe Modell !! 0 !! 0,25 !! 0,45 !! 0,55 !! 0,75 | ||
| Line 79: | Line 91: | ||
| krank || 0,56 || style="background:#B9FFC5 " | TP || style="background:#B9FFC5 " | TP|| style="background:#B9FFC5 " | TP || style="background:#B9FFC5 " | TP || style="background:#FFCBCB| FN | | krank || 0,56 || style="background:#B9FFC5 " | TP || style="background:#B9FFC5 " | TP|| style="background:#B9FFC5 " | TP || style="background:#B9FFC5 " | TP || style="background:#FFCBCB| FN | ||
|- | |- | ||
| gesund || 0, | | gesund || 0,56 || style="background:#FFCBCB| FP || style="background:#FFCBCB| FP || style="background:#FFCBCB| FP || style="background:#FFCBCB| FP || style="background:#B9FFC5 " | TN | ||
|- | |- | ||
| gesund || 0,1 || style="background:#FFCBCB| FP || style="background:#B9FFC5 " | TN || style="background:#B9FFC5 " | TN || style="background:#B9FFC5 " | TN || style="background:#B9FFC5 " | TN | | gesund || 0,1 || style="background:#FFCBCB| FP || style="background:#B9FFC5 " | TN || style="background:#B9FFC5 " | TN || style="background:#B9FFC5 " | TN || style="background:#B9FFC5 " | TN | ||
| Line 87: | Line 99: | ||
| gesund || 0,4 || style="background:#FFCBCB| FP || style="background:#FFCBCB| FP || style="background:#B9FFC5 " | TN || style="background:#B9FFC5 " | TN || style="background:#B9FFC5 " | TN | | gesund || 0,4 || style="background:#FFCBCB| FP || style="background:#FFCBCB| FP || style="background:#B9FFC5 " | TN || style="background:#B9FFC5 " | TN || style="background:#B9FFC5 " | TN | ||
|} | |} | ||
{| class="wikitable" | {| class="wikitable" | ||
|- | |- | ||
! !! style="background:orange|0 !! style="background:yellow|0,25 !! style="background:#3944BC|0,45 !! style="background:red|0,55 !! style="background:green|0,75 | |||
|- | |- | ||
| | | Spezifität || 0 ||1/2 || 3/4 || 3/4 || 1 | ||
|- | |- | ||
| | | Sensitivität || 1 || 3/4 || 3/4 || 1/2 || 0 | ||
|} | |} | ||
Der beste Threshold ist 0,45 | |||
==Area Under the Curve== | |||
[[File:AUC.png|frameless|500px|left]] | |||
<br><br> | |||
*1 = optimal <br> | *1 = optimal <br> | ||
*0 = auch gut, positiv und negativ müssen nur umgedreht werden <br> | *0 = auch gut, positiv und negativ müssen nur umgedreht werden <br> | ||
*0,5 = schlecht, da die Bestimmung des Systems durch Zufall erfolgt | *0,5 = schlecht, da die Bestimmung des Systems durch Zufall erfolgt | ||
Latest revision as of 15:52, 31 January 2021
Biomarker
Medizinische Biomarker
- Ein Biomarker ist ein messbarer Parameter biologischer Prozesse, der prognostische, oder diagnostische Aussagekraft hat und somit ein Indikator für Krankheiten, oder Umwelteinflüsse ist.
- Charakteristische biologische Merkmale, die objektiv gemessen werden können und auf einen normalen biologischen, oder krankhaften Prozess im Körper hinweisen können.
- Es können Zellen, Gene, Genprodukte, Enzyme, oder Hormone sein.
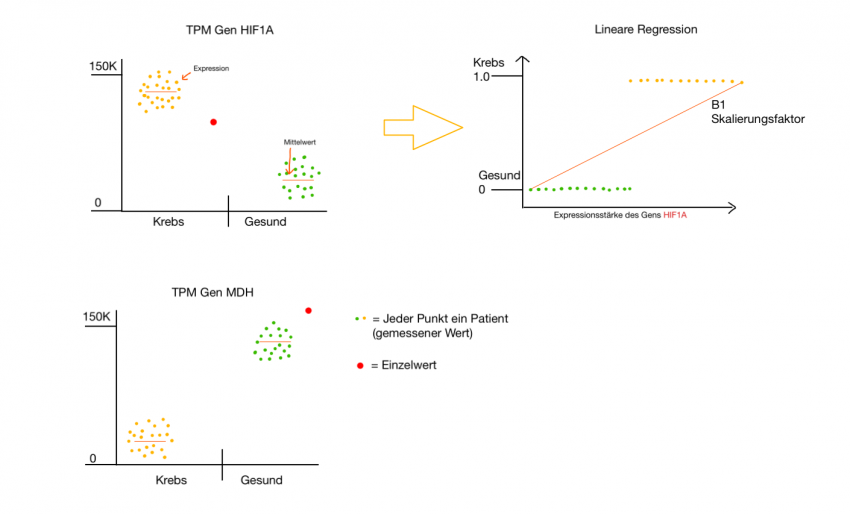 Wie in der Abbildung oben dargestellt, kann über Einzelwerte keine Aussage getroffen werden, ob der Patient krank oder gesund ist.
Wie in der Abbildung oben dargestellt, kann über Einzelwerte keine Aussage getroffen werden, ob der Patient krank oder gesund ist.
→ deswegen bedarf es mehrerer Biomarker
Frage:
Wie kann für einen TPM-Wert vorhergesagt werden, ob es sich um 'Krebs' oder 'Gesund' handelt?
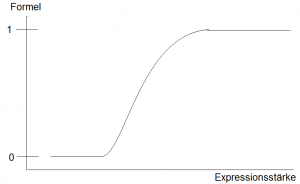
Logistische Funktion
[math]\displaystyle{ y = \beta_1 \cdot TPM_{HIF1\alpha} + \beta_0 }[/math]
Damit wird die Wahrscheinlichkeit, dass jemand z.B. Krebs bekommt durch Gegenwahrscheinlichkeit bestimmt.
[math]\displaystyle{ logit_{(P)} = log \frac{P}{1-P} }[/math]
Der Wertebereich der Logit Funktion ist ideal für lineare Regression
[math]\displaystyle{ log \frac{P(x)}{1-P(x)} = \beta_1 \cdot TPM_{HIF1\alpha} + \beta_0 }[/math]
Biomarkersignatur
Erweiterung für mehrere Gene:
[math]\displaystyle{ P(x) = \frac{1}{e^{-((\beta)_1 \cdot x_1) + ((\beta)_2 \cdot x_1) + .... + (\beta)_n \cdot x_n) + \beta_0)}} }[/math]
Mashine learning
- Ein Modell muss zunächst von einem System erlent werden, dann kann das System eine Aussage treffen, ob z.B. 'krank' oder 'gesund'.
- Parameter [math]\displaystyle{ \beta_1 }[/math] und [math]\displaystyle{ \beta_0 }[/math] können nicht berechnet werden. → Müssen mit Hilfe eines Algorithmus geschätzt werden.
Maximum-Likelihood:
- Schätzung zur Parameterbestimmung.
- Training: Lerndaten sind bekannte Werte zur Aussage 'krank' oder 'gesund'
→ Algorithmus bestimmt einen genauen Wert für [math]\displaystyle{ \beta_1 }[/math], um krank (1) und gesund (0) zu bestimmen.
→ Danach kann mit dem Modell bestimmt werden, ob es gut oder schlecht ist.
Problem: Das System versucht einen optimalen Wert zu finden
→ Overfitting: Zu genaue Beschreibung des Modells.
Frage: Wie gut ist die Qualität des Modells?
Receiver Operation Characteristic (ROC)
Wird genutzt um zu prüfen, ob das Modell gut ist.
→ Wie viele falsch positive, falsch negative, aber auch richtig positive und richtig negative Aussagen trifft das Modell?
| Mensch | ||||
| Krebs | Gesund | |||
| Modellvorhersage | Krebs | TP | FP | |
| Gesund | FN | TN | ||
Beispiel 'Krebs' = positiv, 'geund' = negativ
- TP: True Positive (richtig positiv) → Modell sagt 'Krebs' richtig voraus.
- TN: True Negative (richtig negativ) → Modell sagt 'gesund' richtig voraus.
- FP: False Positive (falsch positiv) → Modell sagt der Patient hat Krebs, obwohl er gesund ist.
- FN: False Negative (falsch negativ) → Modell sagt 'gesund', obwohl der Patient Krebs hat.
Testen auf Sensitivität
Möglichst viele 'Kranke' sollen richtig bestimmt werden.
[math]\displaystyle{ \text{Sensitivität} = \frac{\text{TP}}{\text{TP + FN}} }[/math]
- Bei einem perfekten Modell gibt dieses keine falsch negativen Ergebnisse aus.
- Ausgabe 1 = Perfektes Modell, erkennt alle mit Krebs.
- Ausgabe 0 = Schlechtes Modell, erkennt keine mit Krebs.
Testen auf Spezifität
Möglichst viele 'Gesunde' sollen richtig bestimmt werden.
[math]\displaystyle{ \text{Spezifität} = \frac{\text{TN}}{\text{ FP + TN}} }[/math]
- Bei einem perfekten Modell gibt dieses keine falsch positiven Ergebnisse aus.
- Ausgabe 1 = Perfektes Modell, erkennt alle mit gesund.
- Ausgabe 0 = Schlechtes Modell, erkennt keine gesunden.
- Entweder hohe Sensitivität oder hohe Spezifität. Was besser ist kommt auf die Fragestellung an.
Treshold:
Cutoff-Wert der als Grenzwert festgelegt wird nach dem beurteilt wird, ob der zu bewertende Wert 'krank', oder 'gesund' zugeordnet wird.
Beispiel zur Bestimmung des optimalen Treshold
gesund=0 und krank=1
| Treshold | |||||||
| Zustand | Ausgabe Modell | 0 | 0,25 | 0,45 | 0,55 | 0,75 | |
|---|---|---|---|---|---|---|---|
| krank | 0,5 | TP | TP | TP | FN | FN | |
| krank | 0,6 | TP | TP | TP | TP | FN | |
| krank | 0,2 | TP | FN | FN | FN | FN | |
| krank | 0,56 | TP | TP | TP | TP | FN | |
| gesund | 0,56 | FP | FP | FP | FP | TN | |
| gesund | 0,1 | FP | TN | TN | TN | TN | |
| gesund | 0,2 | FP | TN | TN | TN | TN | |
| gesund | 0,4 | FP | FP | TN | TN | TN | |
| 0 | 0,25 | 0,45 | 0,55 | 0,75 | |
|---|---|---|---|---|---|
| Spezifität | 0 | 1/2 | 3/4 | 3/4 | 1 |
| Sensitivität | 1 | 3/4 | 3/4 | 1/2 | 0 |
Der beste Threshold ist 0,45
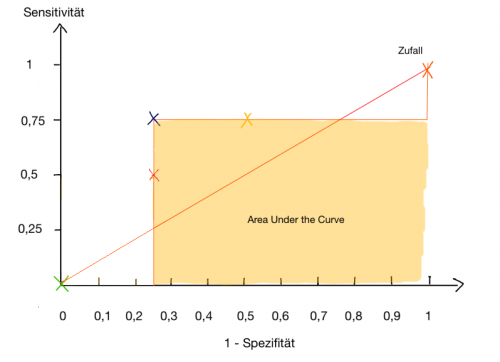
Area Under the Curve
- 1 = optimal
- 0 = auch gut, positiv und negativ müssen nur umgedreht werden
- 0,5 = schlecht, da die Bestimmung des Systems durch Zufall erfolgt