2 Transkriptom RNA Seq 1: Difference between revisions
From Bioinformatik Wiki
(→FASTQ) |
|||
| Line 34: | Line 34: | ||
<big> | <big> | ||
@ | @ Identifier # Sequenz identifier | ||
GATCTT # Sequenz | GATCTT # Sequenz | ||
+ optionale Beschreibung | + # optionale Beschreibung | ||
!'CC'*+*!? # Qualität für jedes Level (Zahlenwert repräsentiert durch [https://de.wikipedia.org/wiki/American_Standard_Code_for_Information_Interchange#ASCII-Tabelle ASCII Tabelle] | !'CC'*+*!? # Qualität für jedes Level (Zahlenwert repräsentiert durch [https://de.wikipedia.org/wiki/American_Standard_Code_for_Information_Interchange#ASCII-Tabelle ASCII Tabelle]) | ||
</big> | </big> | ||
</br> | </br> | ||
Revision as of 19:51, 28 January 2021
Biologische Fragestellung
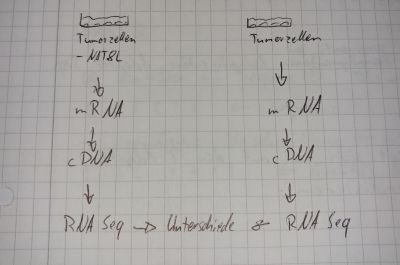
Da NAT8L in euren Prostatakarzinomzellen erhöht exprimiert ist, habt ihr euch entschlossen die Genexpression durch RNAi (RNA Interferenz) zu silencen.
Experiment
- Aus zwei Tumorzellkulturen wird die mRNA extrahiert
- mit Hilfe der reversen Transkriptase wird cDNA transkribiert
- cDNA wird sequenziert
- Sequenzunterschiede können analysiert werden
Sanger Methode
Als Wiederholung:
Next Generation Sequencing (NGS)
Illumina Sequencing (2nd Generation Sequencing)

The DNA attaches to the flow cell via complementary sequences. The strand bends over and attaches to a second oligo forming a bridge. A polymerase synthesizes the reverse strand. The two strands release and straighten. Each forms a new bridge (bridge amplification). The result is a cluster of DNA forward and reverse strands clones.
Ergebnis der Illumina Sequenzierung:
- Länge der reads 50-600bp
- Fehlerrate ca. 0,1%
- humanes Genom kann 30x am Tag sequenziert werden
- Daten werden in FASTQ Format geliefert
FASTQ
Eine FASTQ-Datei ist folgendermaßen aufgebaut:
@ Identifier # Sequenz identifier GATCTT # Sequenz + # optionale Beschreibung !'CC'*+*!? # Qualität für jedes Level (Zahlenwert repräsentiert durch ASCII Tabelle)
Problem
Viele kurze reads, die in einen Zusammenhang gebracht werden müssen!
Sequenzabdeckung
G: Länge der Genomsequenz
N: Anzahl der reads
L: durchschnittliche Länge der reads
C: Coverage (Abdeckung)
[math]\displaystyle{ C= \frac{N*L}{G} }[/math]
Lander-Waterman-Modell
Mathematisches Modell zur Errechnung, durch Sequenzierung, nicht abgedeckter Basenpaare.
P[nicht abgedecktes Bp] = e-c
Beispiele:
c=10 → 1 Gap in 22000 Bp
c=22 → 1 Gap in 3,6*109 Bp
(c meist bei 30)
Assemblierung der reads
| -A | -C | -C | -T | -G | -A | -C | T | -A | -G | -C | -T | -G | -A | -T | -C | -A | -A | -G | -G | - | - | Template | |||
| -G | -A | -T | -C | -A | -A | ||||||||||||||||||||
| -A | -G | -C | -T | -G | -A | ||||||||||||||||||||
| -A | -C | G | -A | -G | -C | -T | - | - | Punktmutation | ||||||||||||||||
| -G | -A | -_ | -C | -A | -A | -G | -G | - | - | Deletion |
![400px][https://de.wikipedia.org/wiki/DNA-Sequenzierung#Didesoxymethode_nach_Sanger](https://upload.wikimedia.org/wikipedia/commons/thumb/d/d9/Didesoxy-Methode.svg/960px-Didesoxy-Methode.svg.png)