5 Transkriptom RNA Seq 2: Difference between revisions
From Bioinformatik Wiki
No edit summary |
No edit summary |
||
| Line 1: | Line 1: | ||
= RNAseq = | = RNAseq = | ||
<br> | <br> | ||
Revision as of 12:58, 19 May 2020
RNAseq
1. Isolierung von mRNA
- nur 1-2% der totalen RNA ist mRNA
- 90% rRNA
- poly(A) Anreicherung
- Abbau von rRNA
2. cDNA Synthese und Library Präparation
- Information über kodierenden Strang kann durch 2-stufige cDNA Synthese und dUTP Nukleotiden konserviert werden.
| A | U | G | U | C | G | A | mRNA | ||
| T | A | C | A | G | C | U | 1. cDNA Strang | ||
| T | A | C | A | G | C | U | |||
| A | U | G | U | C | G | A | 2. cDNA Strang mit dUTPs statt dTTPs |
⇒ Library Präparation, Adapter Legierung
⇒ Abbau des 2. Strangs durch Uracil-DNA Glykosylase
→ PCR/ Amplifizierung / Sequenzierung
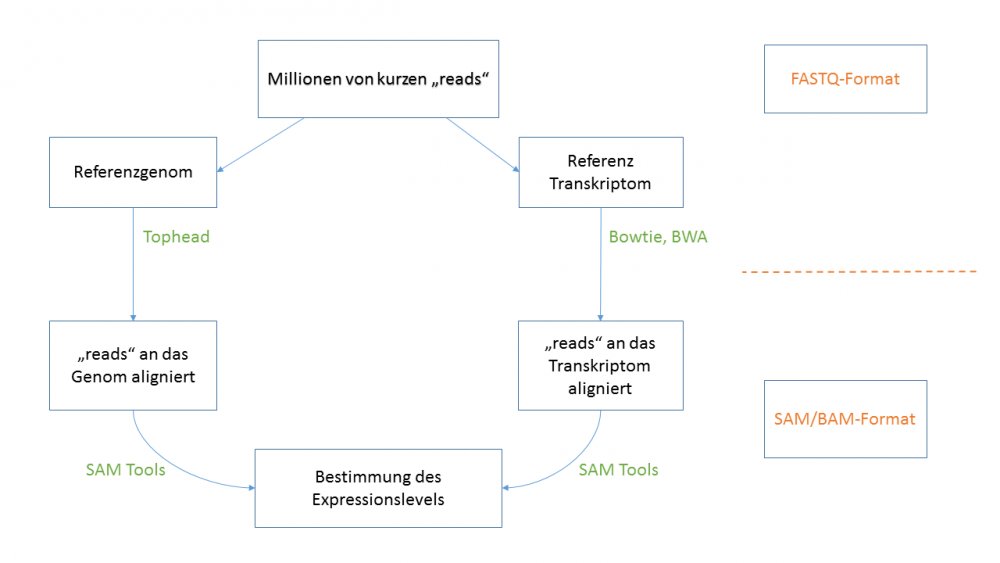
3. Sequenzierung/ Next generation Sequencing
⇒ Sequenziertiefe bestimmt die Empfindlichkeit und die Genauigkeit
⇒ 5 Millionen reads ausreichend für mittel-hoch exprimierte Gene
⇒ 100 Millionen reads für schwach exprimierte Gene
⇒ Wichtig: Anzahl an Replikaten bestimmt statistische Sensitivität für Unterschiede
| Replikate pro Gruppe | |||
| fold change | 3 | 5 | 10 |
| 1,25 | 17% | 25% | 44% |
| 1,5 | 43% | 64% | 91% |
| 2 | 87% | 98% | 100% |
4. Präprozessierung der Rohdaten
⇒ Filtern von Basen mit geringer Qualität
⇒ Trimmen von Adaptersequenzen und PCR-Primersequenzen
Programme: FASTQC, NGSQC, Trimmomatic