3 Alignments: Difference between revisions
From Bioinformatik Wiki
No edit summary |
No edit summary |
||
| Line 61: | Line 61: | ||
M<sub>k,0</sub>=0<br> | M<sub>k,0</sub>=0<br> | ||
M<sub>k,j</sub>=max(0, M<sub>k-1,j</sub>+gp,M<sub>k,j-1</sub>+gp,M<sub>k-1,j-1</sub>+equal(k,j))<br> | M<sub>k,j</sub>=max(0, M<sub>k-1,j</sub>+gp,M<sub>k,j-1</sub>+gp,M<sub>k-1,j-1</sub>+equal(k,j))<br> | ||
[[File:Alignment.png|frameless|700px]] | |||
Revision as of 00:52, 16 May 2019
Auf dieser Seite sind die Themen zusammengeführt, die in Vorlesung 3 am 25.04.2019 behandelt wurden.
Alignments
→ Für ein Alignment werden spezielle Algorithmen benötigt.
Sequenz Alignments
- Ähnlichkeit quantitativ bestimmbar
- Erkennung ähnlicher Domänen
- erfordert das Einfügen von "gaps"
Globales Alignment
- alle Symbole werden berücksichtigt
- Vergleich ähnlicher Sequenzen mit ähnlicher Länge
Beispiel:
| H | A | U | S | K | L | A | U | S | ||
| ↓ | ||||||||||
| H | - | A | U | S | - | H | A | U | S | |
| K | L | A | U | S | K | L | A | U | S | |
| Score 8 | Score 8 | |||||||||
Bewertungsschema: S(a,b)={3 a=b match ;0 a≠b mismatch
| gaps: | S(a,-)=-1 Deletion |
| S(-,b)=-1 Insertion |
Lokales Alignment
- Alignment von Teilsequenzen
- Vergleich zweier sehr unterschiedlicher Sequenzen, die aber gleiche Motive besitzen
- Suche einer Gensequenz im Genom
Beispiel:
| A | T | G | C | A | T | T | A | C |
| C | T | T | T | A |
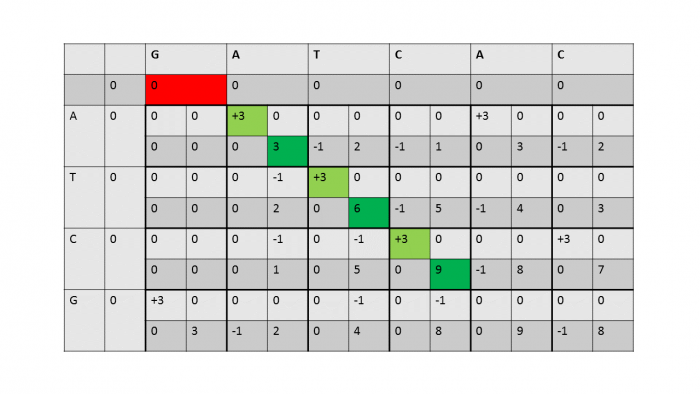
Smith-Waterman Algorithmus
Dynamic programming: "divide and conquer", Aufteilen des Problems in Subprobleme
M0,0=0
M0,j=0
Mk,0=0
Mk,j=max(0, Mk-1,j+gp,Mk,j-1+gp,Mk-1,j-1+equal(k,j))