2 Transkriptom RNA Seq 1
Biologische Fragestellung
Das NAT8L Gen scheint im Metabolismus von Krebszellen eine Rolle zu spielen. Unser Projekt befasst sich mit Lungenkrebs. Daraus folgt die Frage: Ist NAT8L in Lungenkrebszellen erhöht exprimiert ?
Die Frage impliziert weitere Überlegungen z.B. ob NAT8L essentiell für das Wachstum von Krebszellen ist und ob eine Inhibierung der NAT8L Expression die Tumorbildung hemmen würde.
Um die Frage zu beantworten sind folgende drei Schritte notwendig:
Schritt 1:
- Abfragen von Datenbanken (vielleicht wurden bereits ähnliche Experimente durchgeführt)
Schritt 2:
- aus Datenbanken haben wir herausgefunden, dass NAT8L in Lungenkrebszellen erhöht expliziert wird
Schritt 3:
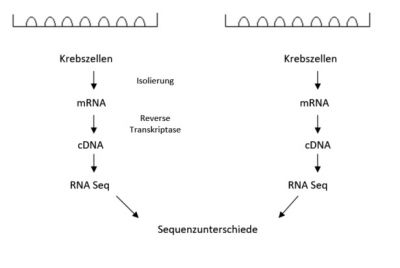
- in einem Experiment soll versucht werden die Genexpression durch RNAi (RNA interference) zu silencen
- aus zwei Tumorzellkulturen wird die mRNA extrahiert
- mit Hilfe der reversen Transkriptase wird cDNA transkribiert
- cDNA wird sequenziert
- Sequenzunterschiede können analysiert werden
Dem Experiment liegt die Transkriptomanalyse zugrunde, welche eine quantitative und qualitative Bestimmung aller Transkriptionsaktivitäten ermöglicht. D.h. die Erfassung aller mRNA-Moleküle, wodurch auf die Aktivität bestimmter Gene unter bestimmten Bedingungen zurückschließen lässt. Eine Trannskriptomanalyse (also die Erfassung der Gesamtheit aller in einer Zelle unter einer bestimmten Bedingung erzeugten mRNA Moleküle) lässt sich auf verschiedene Weisen durchführen. Eine Möglichkeit ist ein DNA-Microarray (s. unten). Eine andere Möglichkeit ist die RNA-Sequenzierung. Zum Ablauf RNA-Sequenzierung vergleiche 5_Transkriptom RNA Seq 2. Für beide Analysemethoden ist jedoch die gleiche sample preparation notwendig.
- mRNA Extraktion aus dem Organismus
- mRNA wird durch Reverse Transkriptase zu cDNA umgeschrieben
→ dann folgt die Expressionsanalyse durch genannte Methoden
Sequenzierung
Für die quantitativen und qualitativen Bestimmungen von Sequenzen bei der Transkriptomanalyse durch die RNA-Sequenzierung müssen, wie der Name schon verrät, die cDNA-Sequenzen sequenziert werden. Hierfür werden die NGS Methoden wie Illumina verwendet.
Next Generation Sequencing (NGS)
Illumina Sequencing (2nd Generation Sequencing)
Ergebnis der Illumina Sequenzierung:
- Länge der reads 50-600bp
- Fehlerrate ca. 0,1%
- humanes Genom kann 30x am Tag sequenziert werden
- Daten werden in FASTQ Format geliefert
FASTQ
Die NGS-Sequenzierergebnisse werden in diesem textbasierten-Format geliefert, dass zur Speicherung von DNA/RNA-Sequenzen dient.
Eine FASTQ-Datei ist folgendermaßen aufgebaut:
@ Identifier # Sequenz identifier GATCTT # Sequenz + # optionale Beschreibung !'CC'*+*!? # Qualität jedes Nukleotids (Zahlenwert repräsentiert durch ASCII Tabelle)
Problem
Viele kurze reads, die in einen Zusammenhang gebracht werden müssen! Um Lücken in der Sequenz zu vermeiden, muss die Sequenziertiefe angepasst werden.
Microarrays
In Zuge dieser Vorlesung wird unter Microarrays die DNA-Chip Microarray-Methode verstanden. Mit dieser molekularbiologischen Methode lässt sich der Grad der Genexpression in einem Organismus von einer großen Anzahl an Genen gleichzeitig messen. Es wird also gemessen, welche Gene unter welchen Bedingungen wie stark aktiv sind. Stellt Damit eine alternative zur RNA-Seq. dar, bei der die Auswertung mit Hilfe von NGS erfolgt.
Ablauf
Auf einer Oberfläche z.B. Glas befinden sich in einer bestimmten Anordnung immobilisierte DNA-Fragmente, so dass die Sequenz der DNA-Fragmente an jeder Position bekannt ist. Diese kurzen Oligonucleotide sind Sequenzabschnitte bekannter Gene des zu untersuchenden Organismus und deshalb komplementär zu cDNA-Sequenzen, dessen qualitative und quantitative Expression überprüft werden soll. Wobei sich nicht nur um ein Oliginukleotid sondern um eine große Anzahl der selben Oligonucleotide auf einen spot handelt. Nach der sample preparation erhält man cDNA. Die einzelsträngige cDNA ist floureszens markiert. Die Markierung kann z.B. durch die Verwendung von bereits floureszenzmarkierten dNTPs während der cDNA-Synthese erfolgen. Die cDNA der Probe wird auf den Chip aufgetragen und hybridisiert mit den komplementären Sequenzen. Nach der Fixierung werden die ungebundenen cDNA-Stränge heruntergewaschen. Überall dort wo dann Floureszenz zu messen ist, hat die cDNA an die komplementäre Oligonucleotid gebunden. Weil bekannt ist an welcher Stelle, welches Gensequenzoligonukleotid immobilisiert ist, kann so darauf geschlossen werden, welche Gene im untersuchten Organsimus exprimiert wurden. Die Intensität der Floureszenz korreliert mit der Anzahl an gebundenen cDNA Strängen pro Spot, wass wiederum mit dem Expressionslevel des Gens korreliert.
Hauptsächlich werden sie genutzt um mehrer Proben gleichzeitig zu Vergleichen. Wobei es sich um den vergleich der Genexpressionsmuster unter versch. Bedingungen handelt.
Man unterscheidet dabei zwei Herangehensweisen:
One-color technique:
Die cDNA von zwei verscheiden Proben werden mit unterschiedlichen Farben markiert. Die Proben werden jeweils auf ein Mikroarray gegeben und hybridisieren dort. Die Auswertung erfolgt indem die Bilder der Microarrays übereinander gelegt werden. Dort wo nur cDNA einer Probe gebunden hat, ist die entsprechende Farbe der Probe zu beobachten, dort wo beide gebunden haben, ist eine Farbe zu beobachten, die sich aus der Mischung beider einzelnen Probenfarben ergibt.
Two-color technique:
Die cDNA von zwei verschiedenen Proben werden mit unterschiedlichen Farben markiert. Die zwei Proben werden dann zusammengemsicht und auf einen DNA Chip gegeben und hybridisieren dort kompetitiv. Dort wo nur cDNA einer Probe gebunden hat, ist die entsprechende Farbe der Probe zu beobachten, dort wo beide gebunden haben, ist eine Farbe zu beobachten, die sich aus der Mischung beider einzelnen Probenfarben ergibt.
Nachteile
Die Microarray-Technologie tritt heutzutage etwas in den Hintergrund, da die Methode einigen Einschränkungen unterliegt. Zum einen löst das Microarray die Sequenz nicht auf die Einzelbase auf, weshalb Mutanten und Splicevarianten oftmals nicht bestimmt werden können. Dies liegt daran, dass auch Sequenzen hybridisieren, die nicht 100% übereinstimmen. Außerdem beruht die Methode auf optischer Messung, was bedeutet, dass ein gewisses Maß an Hintergrundrauschen unvermeidbar ist und eine Messuntergrenze besteht. Somit kann es vorkommen, dass geringe Mengen an bestimmten Sequenzen nicht nachgewiesen werden können. Diese Methode besitzt zudem eine Messobergrenze, denn wenn alle Sonden in einem Spot hybridisiert haben, kann weitere komplementäre cDNA nicht binden und verfällt wenn sie herunter gewaschen wird. Zudem können nur Proben von Organismen verwendet werden, bei denen die Sequenz bereits bekannt ist.
Assembly
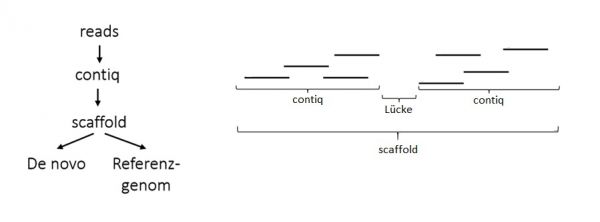
Assembly ist ein Bioinformatisches Verfahren, bei dem überlappende reads zu Contiqs und anschließend zu Scaffolds zusammengesetzt werden. Wenn das Genom bereits bekannt ist können die Scaffolds an ein Referenzgenom aligned werden, wobei man diese Art der Assemblierung als Alignment oder Assemblierung mit Hilfe eines Referenzgenoms bezeichnet. Liegt keine Referenz vor, bezeichnet man sie als de novo Assemblierung.
Contiq: Zusammenhängender Abschnitt einer genomischen Sequenz, die aus überlappenden reads gebildet wird. Zwischen ihnen befinden sich noch Lücken.
Scaffold: Bestehend aus Contiqs und Lücken (auch Supercontiqs genannt); definieren die Reihenfolge, Orientierung und Größe der Lücken zwischen den Contiqs.
Beim NGS wird eine gewünschte Target-Sequenz mithilfe der Shotgun-Methode sequenziert, sodass viele kleine DNA-Fragmente (reads) entstehen. Diese sind vollkommen ungeordnet und in keiner Weise miteinander in Bezug gesetzt. Durch das Assembly wird die eigentliche Sequenz aus den erhaltenen reads rekonstruiert und zusammengesetzt. Da NGS heutzutage vollem bei größeren Sequenzierungen hauptsächlich angewendet werden, ist immer eine Assemblierung des reads notwendig.
Lander-Waterman-Modell
- stellt die statistische Wahrscheinlichkeit für das Auftreten von gaps dar
- dient somit der Errechnung nicht abgedeckter Basenpaare bei einer Sequenzierung
P[nicht abgedecktes Bp] = e-c
- P gibt die Anzahl an Basenpaaren an nach denen statistisch ein gap erwartet wird
- dabei ist C die Coverage, also die Anzahl an reads an einer beliebigen Stelle der Sequenz, die diese Stelle abdecken
- rechnet man mit der gegeben Formel für P, erhält man erstmal die Wahrscheinlichkeit für das Auftreten eines gaps, bei einer bestimmten Coverage
- um die tatsächliche Anzahl an bp zu bekommen nach der statistisch ein gap auftritt, ist es notwenndig
[math]\displaystyle{ \frac{1}{P} }[/math] zu teilen
G: Länge der Genomsequenz
N: Anzahl der reads
L: durchschnittliche Länge der reads
C: Coverage (Abdeckung)
[math]\displaystyle{ C= \frac{N*L}{G} }[/math]
Beispiele:
C=10 → 1 Gap in 22000 Bp
C=22 → 1 Gap in 3,6*109 Bp
C=30 → So tief, dass quasi alles überdeckt wird