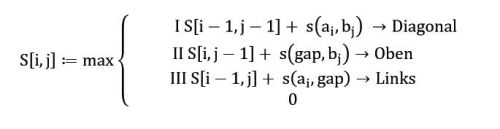3.Smith-Waterman
Aufgabe 1: Definitionen
a: Definiere die folgenden Begriffe:
- Alignment: Methode, bei der verschiedene Sequenzen optimal aneinander ausgerichtet werden, um übereinstimmende Teile untereinander sichtbar zu machen. Die kann entweder zwischen einem Read und einem Referenzgenom passieren oder zwischen zwei Reads im Zuge der RNAseq.
- Dynamic Programming: Algorithmus zum Lösen von informatischen Problemen. Dabei wird ein Problem in Teilprobleme unterteilt, die nach und nach gelöst und gespeichert werden. Die kann auf Assemblies angewandt werden, indem man die reads nach und nach aneinanderfügt, anstatt dies in einem einzigen Programmschritt durchzuführen. Dabei wird stets ein Paar mit maximalem Match verbunden bevor das nächste Paar verwendet wird.
- lokales Alignment: Alignment von zwei (Teil)-Sequenzen, wird mit dem Smith-Waterman-Algorithmus durchgeführt. Zur Berechnung müssen die Teilsequenzen gefunden werden, die den höchsten Alignmentscore besitzen.
- Gap penalty: Bewertung von Lücken, die in ein Alignment eingefügt wurden. Als Folge ein Abzug im Alignmentscore, wenn bei einem lokalen Alignment eine Base ungepaart ist (Das Beispiel hat an Position 3 ein Gap und somit eine Gap penalty.
AATTGGCC TT-AACCGG
- Algorithmus: Verarbeitungsvorschfrift, die aus einer endlichen Folge von eindeutig ausführbaren Anweisungen besteht, mit der man eine Vielzahl gleichartiger Probleme lösen kann. Alternative Definition: Handlungsanweisung zur Lösung eines Problems, besteht aus definierten, sequentiellen Abschnitten.
- Fasta-Format: textbasiertes Format in dem DNA, RNA und Proteinsequenzen gespeichert werden. Besteht aus 2-3 Zeilen pro Sequenz. Die erste Zeile besteht aus einem Header, der mit > beginnt und Namen und Beschreibung der Sequenz beinhaltet. Danach folgt eine optinale Kommentarzeile, gefolgt von der Zeile mit der eigentlichen Sequenz. Mehrere Sequenzen können in einer Datei gespeichert werden, wenn man einen neuen Header für jede Sequenz beginnt.
Beispiel:
>Probesequenz_1_20190502_ABC AATTAAGCATAAATAGGCTAGCTAAGCTAGCCA >Probesequenz_2_20190502_ABC GGGATTCGACCGATCGAAGCTTAGCGAACGAGA |
b: Welche grundsätzlichen Arten von Alignment gibt es?
Lokales Alignment: Alignment von unterschiedlichen (Teil)-Sequenzen, normalerweise unter der Verwendung des Smith-Waterman-Algorithmuses. Wird mit längeren Teilsequenzen immer aufwändiger.
Globales Alignment: Volle Sequenzen werden miteinander alignt. Wird oftmals mit dem Needleman-Wunsch Algorithmus bearbeitet.
Globales Alignment: Vergleich von Gesamtsequenzen Lokales Alignment: Vergleich von Teilsequenzen
Aufgabe 2: Smith-Waterman
a: Erkläre welche Funktion der Smith-Waterman-Algorithmus hat und wie er funktioniert.
Der Smith-Waterman Algorithmus wird verwendet um lokale Alignments zu finden.
Berechnet wird es mit folgender Matrix:
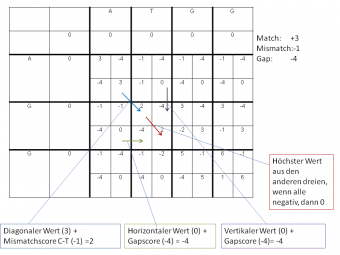
Erklärt bedeutet dies, das eine Matrix aufgespannt wird mit der Größe Länge von Sequenz A x Länge der Sequenz B, so repräsentiert jede Zelle in der Matrix eine Kombination von einer Base aus Sequenz A und einer Base aus Sequenz B und kann somit als Match oder Mismatch identifiziert werden. Eine Zahlenbewertung (Score) für Match, Mismatch und Gap muss vorgegeben werden. Für jede Zelle der Matrix wird ein Wert berechnet, angefangen oben links und beendet unten rechts in der Matrix. Der Wert für eine bestimmte Zelle ist der höchste Wert aus den folgenden 4:
- Der höchste Wert für die Zelle links oben diagonal (oder 0 falls dies außerhalb der Matrix liegt) plus den Score der Zelle (Match oder Mismatch).
- Der höchste Wert aus der Zelle links daneben plus den Score für einen Gap.
- Der höchste Wert aus Zelle oberhalb plus den Score für einen Gap.
- Null
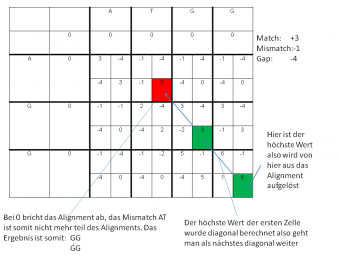
Die Matrix wird berechnet bis alle Werte bestimmt sind. Dann wird der höchste Wert der gesamten Matrix gesucht. Von diesem Punkt beginnt das Alignment. Von dort aus wird rückwärts die Sequenz bestimmt, indem die Rechnung rückwärts verfolgt wird. Der nächste Sequenzteil ist dann jeweils immer die Zelle von der aus man den höchsten Wert berechnet hat. Sobald man jedoch nach links oder nach oben geht anstatt diagonal zu gehen, muss man beachten dass man hierbei ein Gap in das Alignment einbaut und dies auch so aufschreiben muss.
Zuerst berechnet man jede Zelle der Matrix, dann findet man das Aligment
b: Wo in der Smith-Waterman-Matrix beginnt und endet das optimale Alignment?
Das optimale Alignment beginnt bei den höchsten Wert, folgt dann dem Berechnungsstrang bis das erste Mal auf eine 0 getroffen wird. Dort bricht das Alignment ab.
c: Welche Alignment-Art wird durch den Smith-Waterman Algorithmus bestimmt?
Mit dem Algorithmus wird ein lokales Alignment bestimmt.
Aufgabe 3: lokales Alignment
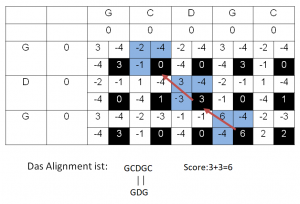
Führe ein Alignment der folgenden Sequenzen durch: GCDGC GDG
Verwende den Ähnlichkeitsscore S: Match = 3 | Mismatch = -2 | Gap = -4
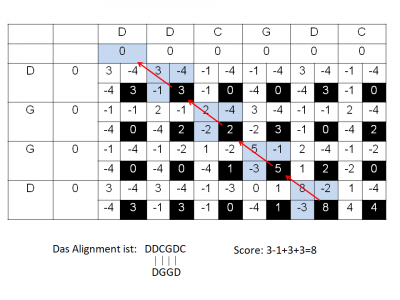
Berechne für die beiden Sequenzen ein optimales lokales Alignment nach Smith-Waterman mit den gleichen Bedingungen. Welches optimale lokale Alignment ergibt sich? Rechts ist die Lösungsmatrix dargestellt, der Score des Alignments ist 6.
Aufgabe 4: Alignment
Während deiner Bachelorarbeit sollst du ein Protein, das du gerade versuchst zu charakterisieren, in einen groben Kontext bringen. Leider weißt du noch nicht viel über das Protein. Daher ist dein erster Schritt, gemeinsame Domänen zwischen den Proteinen zu finden. Für das Protein hat sich folgende Sequenz ergeben: DDCGDC
Durch einen Abgleich der Sequenz mit einer Datenbank kann die Sequenz mit anderen Sequenzen verglichen und ein optimales Alignment gefunden werden. Wieso eignet sich hier der Smith-Waterman Algorithmus? Führe ein Alignment zwischen der oben ermittelten Sequenz und der folgenden durch: DGGD
Ähnlichkeitsscore S: match = 3 | mismatch = -1 | gap penalty = -4
Smith-Waterman nutzt man für lokale Alignments, hier ist dies nützlich da wir Ausschnitte aus zwei funktionell ähnlichen Domänen vergleichen.
Aufgabe 5: Lander-Waterman-Modell
a: Was sagt das Lander-Waterman-Modell aus? Benne die darauf beruhende Formel sowie ihre Bestandteile.
Mit dem Lander-Waterman-Modell kann man berechnen wie viele Gaps in einem Alignment zu erwarten sind.
Die Formel zur Berechung lautet: P(nicht abgedecktes bp)= [math]\displaystyle{ e^{-C} }[/math] wobei [math]\displaystyle{ C= \frac{N*L}{G} }[/math]
Hierbei ist C die Coverage, die sich aus der Anzahl der reads N, der durchschnittlichen readlänge L und der Templatesequenz G berechnet.
b: Suche im Internet die Länge des menschlichen Genoms heraus. Verwende dafür folgende Internetseite: https://www.ncbi.nlm.nih.gov/
Wie viele Reads müssen sequenziert werden, um das humane Genom mit einer Coverage von 30 abzudecken? Gehe hierfür von einer durchschnittlichen Read Länge von 600 bp aus.
Je nach gefundenem Wert für die Genomlänge, die ja nach Genotyp variiert hat man leicht unterschiedliche Werte.
Coverage [math]\displaystyle{ C = 30 = \frac{N*600}{3257320000} }[/math]
[math]\displaystyle{
N = 162866000
}[/math]
c: Wie hoch ist die Häufigkeit nicht abgedeckter bp bei einer Coverage von 40?
Hier können wir das Lander-Waterman-Modell anwenden: [math]\displaystyle{ P=e^{-C} = e^{-40} = 4,24835*10^{-18} }[/math]
d: Wie müsste die Anzahl der Reads gewählt werden, sodass eine möglichst komplette
Übersicht über die Genomsequenz erhalten wird?
Generell gilt, dass je mehr reads desto geringer die Wahrscheinlichkeit dass Gaps autreten. Jedoch verlängert sich damit die Dauer der Sequenzierung auch. Man kann die Lander-Waterman-Formel umstellen um zu berechnen welche Coverage man braucht um nur ein Contig zu haben
[math]\displaystyle{ -C=\ln(\frac{1}{3257320000})=-21,904 }[/math]
Es würde also eine Coverage von über 21 gebraucht werden um statistisch gesehen keine Gaps mehr zu erwarten.